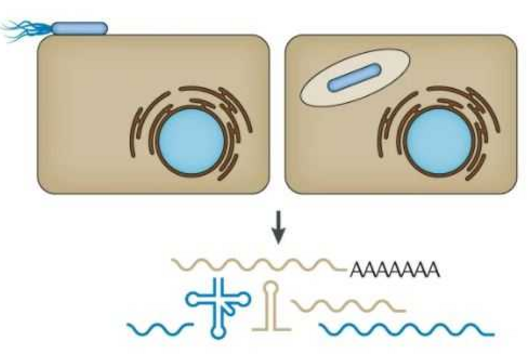
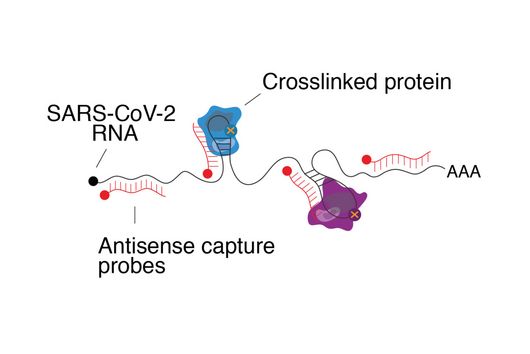
Um zu verstehen, welche Gene in Krankheitserregern aktiv sind, analysieren Infektionsforscher:innen deren Ribonukleinsäuren (RNA). Denn nur von den aktiven Genen einer Zelle werden RNA-Kopien erstellt, damit diese bestimmte genetische Funktionen ausführen können.
Die Gesamtheit der RNA-Moleküle einer Zelle oder eines Gewebes während eines bestimmten Entwicklungszustandes ist also das Produkt derjenigen Gensequenzen, die gerade „angeschaltet“ sind. Dieses RNA-Profil einer Zelle wird auch als Transkriptom bezeichnet und ist Gegenstand der Forschung: Mit dem Ziel, neue therapeutische Ansätze zu entwickeln, wollen Wissenschaftler:innen im Detail verstehen, wie Infektionen verlaufen und welche molekularen Prozesse dabei eine Rolle spielen.
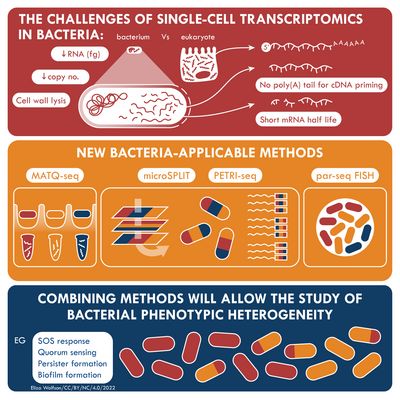
Die Transkriptomanalyse einzelner Zellen mittels Einzelzell-RNA-Sequenzierung (scRNA-seq) ist bei eukaryontischen Geweben – also den Zellen von Tieren, Pflanzen und Pilzen – bereits eine Routinemethode. Das Transkriptom von Bakterienzellen zu entschlüsseln, ist jedoch ungleich schwieriger, etwa weil hier der RNA-Gehalt geringer ist oder die Moleküle instabiler sind (mRNA).
Dank jüngster methodologischer Durchbrüche ist inzwischen auch bakterielles scRNA-seq möglich. In einem Überblicksartikel zum Thema, der kürzlich in dem Fachmagazin microLife erschienen ist, diskutieren Christina Homberger, Lars Barquist und Jörg Vogel die neuen Ansätze und zeigen auf, welche Fortschritte sie ermöglichen und welche technischen Herausforderungen noch auf Lösung warten.

![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/2/a/csm_2024_05_Infection___Immunity_Poster_Webdatei_c3325f947c.png)









![[Translate to German:]](/fileadmin/HIRI/__processed__/3/3/csm_2022_07_BEISEL_Chase_2_cropped_Low_a7a341cf03.jpg)




































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/2/8/csm_Collage_WUE_9fa1cd516c.jpg)










![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/0/8/csm_05_Saliba_02_c_HIRI_Luisa_Macharowsky_ohne_Lacoste_low_zugeschnitten_2_01_9822d73a3f.jpg)





![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/c/csm_HIRI-Grundstseinlegung_Foto__c_Mario_Schmitt_79ad64af6d.jpg)













![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/2/csm_2023_04_Smyth_Paper_c_HIRI_Luisa_Macharowsky_2_low_84f993d581.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/e/2/csm_2022_11_HIRI_City_of_Wuerzburg_Child_Care_c_HIRI_Luisa_Macharowsky_2_low_6445b253eb.jpg)
![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/3/9/csm_HIRI_key_visual_CHurtig_606d992d74.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/f/0/csm_Ahsen_blue2_6e28a4ee6b.jpg)












![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/5/csm_2022_02_Marco_Olguin_HIRI-Luisa_Macharowsky_04_low_new_3dc837191c.jpg)


![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/3/c/csm_Joerg_Vogel_Portrait_-_photocredit_Mario_Schmitt__c__HIRI_5013b9643b.jpg)



![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/6/csm_RNA_Delivery_1902376336.jpg)
![[Translate to Deutsch:]](/fileadmin/HIRI/__processed__/7/c/csm_Caliskan_Kibe_Pekarek_105c7d1860.jpg)
![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/c/1/csm_N-Caliskan_M-Zimmer_Zoom_c93c493293.jpg)