Prof. Mathias Munschauer, Ehemaliger Gruppenleiter
LncRNA und Infektionsbiologie
Von 2019 bis 2023 konzentrierte sich eine Forschungsgruppe unter der Leitung von Mathias Munschauer am Helmholtz-Institut Würzburg (HIRI) auf lncRNA und Infektionsbiologie. Munschauer hat inzwischen eine W3-Professur für „Molekulare Virologie” an der Universität Heidelberg inne und ist weiterhin eng mit der Forschung am HIRI verbunden.
Unsere Forschung
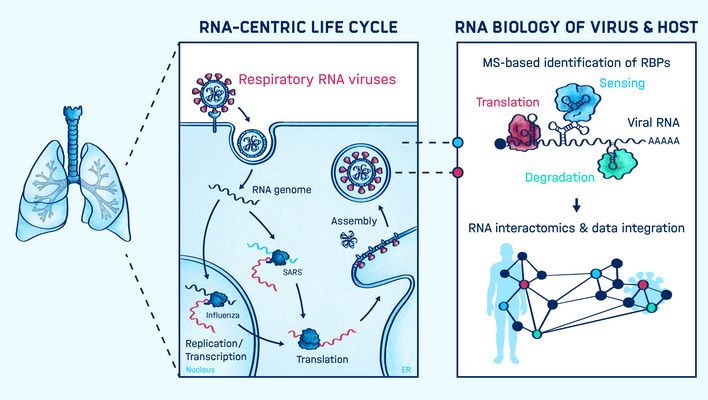
Das Wissen darüber, welche Bestandteile in Wirtszellen an die virale RNA binden und diese während des Infektionsprozesses regulieren, ist entscheidend, um zu verstehen, wie Viren eine Wirtszelle kapern, wie sie Wirtsprozesse unterwandern und wie sie der angeborenen Immunabwehr entgehen können. Einblicke in die zugrunde liegenden molekularen Interaktionen und Mechanismen tragen zur Entwicklung neuartiger RNA-basierter Therapien bei.
Die Arbeitsgruppe von Mathias Munschauer arbeitet an der Erstellung einer Übersichtskarte der funktionell wichtigsten RNA-Protein-Interaktionen für spezifische Typen von RNA, von langen nicht-kodierenden RNAs (lncRNAs) bis hin zu viralen RNA-Genomen.
Bei seiner Arbeit kombiniert das Team von Mathias Munschauer Technologien aus den Bereichen Biochemie, Genomik, Molekularbiologie und Bioinformatik. RNA-Antisense-Aufreinigung und Massenspektrometrie (RAP-MS) werden eingesetzt, um Momentaufnahmen von RNA-Protein-Interaktionen zu erstellen. Diese können anschließend mit Technologien wie RNA-seq, Ribo-seq und CLIP-seq funktionell seziert werden. Das übergeordnete Ziel der Arbeitsgruppe ist es, eine Karte der funktionell wichtigen RNA-Protein-Interaktionen während Infektionen zu erstellen, um die Entwicklung neuer wirtsgerichteter Therapeutika zu unterstützen.
Team-Mitglieder

Prof. Mathias Munschauer, Ehemaliger Gruppenleiter
Gruppenleiter

Lina Günter
Doktorandin

Simone Werner
Technische Assistentin
Forschungsprojekte
Alumni
Jens Aydin, Doktorand • Alexander Gabel, Postdoc • Sabina Ganskih, Doktorandin • Nora Schmidt, Postdoc • Ameya Sinha, Postdoc • Yuanjie Wei, Doktorandin • Sebastian Zielinski, Doktorand
Publikationen
2024
Nucleolar detention of NONO shields DNA double-strand breaks from aberrant transcripts
Trifault B, Mamontova V, Cossa G, Ganskih S, Wei Y, Hofstetter J, Bhandare P, Baluapuri A, Nieto B, Solvie D, …, Munschauer M, Burger K (2024)
Nucleic Acids Research 52 (6): 3050-3068
SHIFTR enables the unbiased identification of proteins bound to specific RNA regions in live cells
Aydin J, Gabel A, Zielinski S, Ganskih S, Schmidt N, Hartigan CR, Schenone M, Carr SA, Munschauer M (2024)
Nucleic Acids Research 52 (5): e26
SARS-CoV-2 antigen rapid detection tests: test performance during the COVID-19 pandemic and the impact of COVID-19 vaccination
Wagenhäuser I, Knies K, Pscheidl T, Eisenmann M, Flemming S, Petri N, McDonogh M, Scherzad A, Zeller D, Gesierich A, …, Gabel A, Krone M (2024)
EBioMedicine 109: 105394
SOX2 interacts with hnRNPK to modulate alternative splicing in mouse embryonic stem cells
Huang Y, Liu Y, Pu M, Zhang Y, Cao Q, Li S, Wei Y, Hou L (2024)
Cell & Bioscience 14 (1): 102
Activated rate-response is associated with increased mortality risk in cardiac device carriers with acute heart failure
Huttelmaier MT, Münsterer S, Morbach C, Sahiti F, Scholz N, Albert J, Gabel A, Angermann C, Ertl G, Frantz S, Störk S, Fischer TH (2024)
PLOS One 19 (4): e0302321
2023
SND1 binds SARS-CoV-2 negative-sense RNA and promotes viral RNA synthesis through NSP9
Schmidt N, Ganskih S, Wei Y, Gabel A, Zielinski S, Keshishian H, Lareau CA, Zimmermann L, Makroczyova J, Pearce C, …, Erhard F, Munschauer M (2023)
Cell 186 (22): 4834-4850.e23
An RNA modification enzyme directly senses reactive oxygen species for translational regulation in Enterococcus faecalis
Lee WL, Sinha A, Lam LN, Loo HL, Liang J, Ho P, Cui L, Chan CSC, Begley T, Kline KA, Dedon P (2023)
Nature Communications 14 (1): 4093
Lab-scale siRNA and mRNA LNP manufacturing by various microfluidic mixing techniques – an evaluation of particle properties and efficiency
Jürgens DC, Deßloch L, Porras-Gonzalez D, Winkeljann J, Zielinski S, Munschauer M, Hörner AL, Burgstaller G, Winkeljann B, Merkel OM (2023)
OpenNano 12 (1): 100161
2022
Congenital anemia reveals distinct targeting mechanisms for master transcription factor GATA1
Ludwig LS, Lareau CA, Bao EL, Liu N, Utsugisawa T, Tseng AM, Myers SA, Verboon JM, Ulirsch JC, Luo W, …, Kanno H, Sankaran VG (2022)
Blood 139 (16): 2534-2546
Protective immune trajectories in early viral containment of non-pneumonic SARS-CoV-2 infection
Pekayvaz K, Leunig A, Kaiser R, Joppich M, Brambs S, Janjic A, Popp O, Nixdorf D, Fumagalli V, Schmidt N, …, Stark K, Nicolai L (2022)
Nature Communications 13 (1): 1018
2021
BRD9 is a druggable component of interferon-stimulated gene expression and antiviral activity
Börold J, Eletto D, Busnadiego I, Mair NK, Moritz E, Schiefer S, Schmidt N, Petric PP, Wong WW, Schwemmle M, Hale BG (2021)
EMBO Reports 22 (10): e52823
Atlas der SARS-CoV-2-RNA-Protein-Interaktionen in infizierten Zellen
Schmidt N, Munschauer M (2021)
BIOspektrum 27 (4): 376-379
The Zinc Finger Antiviral Protein ZAP Restricts Human Cytomegalovirus and Selectively Binds and Destabilizes Viral UL4/UL5 Transcripts
Gonzalez-Perez AC, Stempel M, Wyler E, Urban C, Piras A, Hennig T, Ganskih S, Wei Y, Heim A, Landthaler M, …, Erhard F, Brinkmann MM (2021)
mBio 12 (3): e02683-20
2020
Control of human hemoglobin switching by LIN28B-mediated regulation of BCL11A translation
Basak A, Munschauer M, Lareau CA, Montbleau KE, Ulirsch JC, Hartigan CR, Schenone M, Lian J, Wang Y, Huang Y, …, Lander ES, Sankaran VG (2020)
Nature Genetics 52 (2): 138-145
The lncRNA lincNMR regulates nucleotide metabolism via a YBX1 - RRM2 axis in cancer
Gandhi M, Groß M, Holler JM, Coggins SA, Patil N, Leupold JH, Munschauer M, Schenone M, Hartigan CR, Allgayer H, Kim B, Diederichs S (2020)
Nature Communications 11: 3214
The SARS-CoV-2 RNA-protein interactome in infected human cells
Schmidt N, Lareau CA, Keshishian H, Ganskih S, Schneider C, Hennig T, Melanson R, Werner S, Wei Y, Zimmer M, …, Bodem J, Munschauer M (2020)
Nature Microbiology 6 (3): 339-353
2019
Context-specific regulation of cell survival by a miRNA-controlled BIM rheostat
Labi V, Peng S, Klironomos F, Munschauer M, Kastelic N, Chakraborty T, Schoeler K, Derudder E, Martella M, Mastrobuoni G, …, Rajewsky N, Rajewsky K (2019)
Genes & Development 33 (23-24): 1673-1687
2018
Ribosome Levels Selectively Regulate Translation and Lineage Commitment in Human Hematopoiesis
Khajuria RK, Munschauer M, Ulirsch JC, Fiorini C, Ludwig LS, McFarland SK, Abdulhay NJ, Specht H, Keshishian H, Mani DR, …, Carr SA, Sankaran VG (2018)
Cell 173 (1): 90-103.e19
The NORAD lncRNA assembles a topoisomerase complex critical for genome stability
Munschauer M, Nguyen CT, Sirokman K, Hartigan CR, Hogstrom L, Engreitz JM, Ulirsch JC, Fulco CP, Subramanian V, Chen J, …, Carr SA, Lander ES (2018)
Nature 561 (7721): 132-136
Nuclear lncRNA stabilization in the host response to bacterial infection
Munschauer M, Vogel J (2018)
The EMBO Journal 37 (13): e99875
New insights into the cellular temporal response to proteostatic stress
Rendleman J, Cheng Z, Maity S, Kastelic N, Munschauer M, Allgoewer K, Teo G, Zhang YB, Lei A, Parker B, …, Choi H, Vogel C (2018)
eLife 7: e39054
2017
Developmentally-faithful and effective human erythropoiesis in immunodeficient and Kit mutant mice
Fiorini C, Abdulhay NJ, McFarland SK, Munschauer M, Ulirsch JC, Chiarle R, Sankaran VG (2017)
American Journal of Hematology 92 (9): E513-E519
2016
Systematic mapping of functional enhancer-promoter connections with CRISPR interference
Fulco CP, Munschauer M, Anyoha R, Munson G, Grossman SR, Perez EM, Kane M, Cleary B, Lander ES, Engreitz JM (2016)
Science 354 (6313): 769-773
2015
Comprehensive Protein Interactome Analysis of a Key RNA Helicase: Detection of Novel Stress Granule Proteins
Bish R, Cuevas-Polo N, Cheng Z, Hambardzumyan D, Munschauer M, Landthaler M, Vogel C (2015)
Biomolecules 5 (3): 1441-66
2014
MOV10 Is a 5' to 3' RNA helicase contributing to UPF1 mRNA target degradation by translocation along 3' UTRs
Gregersen LH, Schueler M, Munschauer M, Mastrobuoni G, Chen W, Kempa S, Dieterich C, Landthaler M (2014)
Molecular Cell 54 (4): 573-85
Differential protein occupancy profiling of the mRNA transcriptome
Schueler M, Munschauer M, Gregersen LH, Finzel A, Loewer A, Chen W, Landthaler M, Dieterich C (2014)
Genome Biology 15 (1): R15
High-resolution profiling of protein occupancy on polyadenylated RNA transcripts
Munschauer M, Schueler M, Dieterich C, Landthaler M (2014)
Methods 65 (3): 302-9
2013
Circular RNAs are a large class of animal RNAs with regulatory potency
Memczak S, Jens M, Elefsinioti A, Torti F, Krueger J, Rybak A, Maier L, Mackowiak SD, Gregersen LH, Munschauer M, …, Le Noble F, Rajewsky N (2013)
Nature 495 (7441): 333-8
Identification of LIN28B-bound mRNAs reveals features of target recognition and regulation
Graf R, Munschauer M, Mastrobuoni G, Mayr F, Heinemann U, Kempa S, Rajewsky N, Landthaler M (2013)
RNA Biology 10 (7): 1146-59
2012
The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts
Baltz AG, Munschauer M, Schwanhäusser B, Vasile A, Murakawa Y, Schueler M, Youngs N, Penfold-Brown D, Drew K, Milek M, …, Dieterich C, Landthaler M (2012)
Molecular Cell 46 (5): 674-90
FMRP targets distinct mRNA sequence elements to regulate protein expression
Ascano M, Mukherjee N, Bandaru P, Miller JB, Nusbaum JD, Corcoran DL, Langlois C, Munschauer M, Dewell S, Hafner M, …, Ohler U, Tuschl T (2012)
Nature 492 (7429): 382-6
2010
Transcriptome-wide identification of RNA-binding protein and microRNA target sites by PAR-CLIP
Hafner M, Landthaler M, Burger L, Khorshid M, Hausser J, Berninger P, Rothballer A, Ascano M, Jungkamp A, Munschauer M, …, Zavolan M, Tuschl T (2010)
Cell 141 (1): 129-41
PAR-CliP--a method to identify transcriptome-wide the binding sites of RNA binding proteins
Hafner M, Landthaler M, Burger L, Khorshid M, Hausser J, Berninger P, Rothballer A, Ascano M, Jungkamp A, Munschauer M, …, Zavolan M, Tuschl T (2010)
Journal of Visualized Experiments (41): pii: 2034