Kontakt
Die HIRI-Pressestelle vermittelt Themenexperten, organisiert Dreharbeiten und Interviews und steht für Medienanfragen gern zur Verfügung.

Glossar
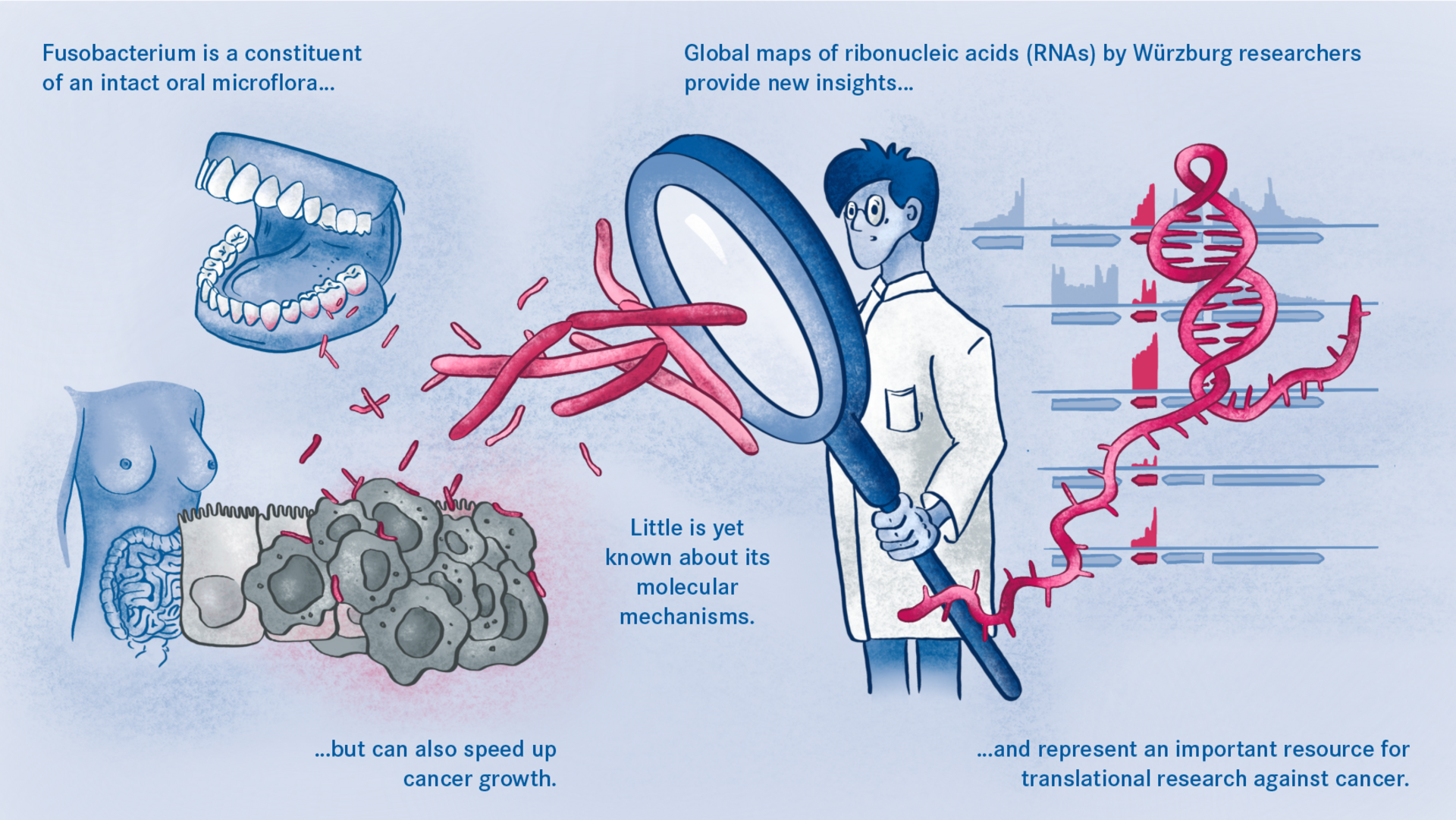
Das HIRI betreibt RNA-basierte Infektionsforschung. Doch was genau ist das eigentlich: RNA? Oder was zum Beispiel ist mRNA, die in einigen der COVID-19-Impfstoffe zum Einsatz kommt?
Diese und viele weitere Fachbegriffe aus unserer Forschung werden in unserem Glossar weiterführend erklärt.
Wenn Sie mehr über unsere Forschung und unsere Wissenschaftler:innen erfahren möchten, klicken Sie bitte auch hier:
Antimikrobielle Wirkstoffe/Antiinfektiva
Antimikrobielle Mittel stoppen oder verlangsamen die Ausbreitung von Mikroorganismen. Neben Antiseptika und Bioziden sind Antiinfektiva eine Klasse antimikrobieller Mittel. Genauer bezeichnet der Begriff Antiinfektiva eine Gruppe von Medikamenten, die Infektionen durch Bakterien, Viren und Pilze verhindern oder behandeln sollen. Die Gruppe umfasst daher antibakterielle, antivirale und antimykotische Medikamente. Ein bekanntes Beispiel für ein antibakterielles Medikament ist Penicillin.
Antisense-Oligonukleotide/RNA-Antibiotika
Antisense-Oligonukleotide sind kleine, synthetische, einzelsträngige Nukleinsäuren, die auf bestimmte Ziel-RNAs ausgerichtet werden können. Diese Moleküle können die Ziel-RNA verändern und die Expression von Proteinen reduzieren, wiederherstellen oder modifizieren, woraus sich ein großes therapeutisches Potenzial ergibt. Bei entsprechender Programmierung können Antisense-Oligonukleotide als hochspezifische „RNA-Antibiotika“ eingesetzt werden. Auf diese Weise könnten sie eine Alternative zu gewöhnlichen Antibiotika bei der Bekämpfung resistenter Krankheitserreger darstellen.
Bayessche Statistik
Bayessche Statistik ist ein Zweig der Statistik, der Wahrscheinlichkeit als Grad persönlicher Überzeugung versteht.
Zellfreie Transkriptions-Translations-Systeme
Zellfreie Transkriptions-Translations-Systeme sind Zell-Lysate, die zur Transkription und zur Translation fähig sind, wenn man ihnen Genkonstrukte hinzufügt. Diese Systeme, meist TXTL genannt, werden für schnelle biochemische Tests in kleinen Volumina verwendet. Sie brauchen keine aufwändigen Verfahren wie Proteinreinigung oder Zellkultivierung, was die Forschungsarbeit erheblich beschleunigt.
CLIP-Seq
CLIP-Seq steht für cross-linking immunoprecipitation-high-throughput sequencing (Quervernetzung und Immunpräzipitation mit Sequenzierung im Hochdurchsatz). Es handelt sich um eine Methode, mit der festgestellt werden kann, welche RNA-Typen miteinander oder mit bestimmten an RNA bindenden Proteinen interagieren.
Kommensale
Kommensale sind Bakterien, die Mikrobiotope eines Menschen oder Tieres besiedeln, ohne gefährlich oder schädlich zu sein.
CRISPR
Die Abkürzung CRISPR steht für Clustered Regularly Interspaced Short Palindromic Repeats (gruppierte kurze Wiederholungen mit regelmäßigen Abständen). Sie bezeichnet eine bestimmte Gruppe sich wiederholender Sequenzen im genetischen Code bestimmter prokaryotischer Organismen wie Bakterien und Archaeen. Diese Gensequenzen sind mit dem Immunsystem der Zelle verknüpft.
CRISPR-Cas
CRISPR-Cas ist ein prokaryotisches Immunsystem, das Wissenschaftler zur Entwicklung gentechnischer Werkzeuge nutzbar gemacht haben. CRISPR-Cas-Technologie besteht aus zwei Komponenten, die die Editierung spezifischer DNA-Segmente von Tieren, Pflanzen und Mikroorganismen ermöglichen: Eine guide-RNA, mit der die gewünschte DNA-Region angesteuert wird, und Cas, eine von der guide-RNA geleitete Nuklease, die die DNA wie eine Schere schneidet. Sobald die DNA geschnitten wurde, lässt sich der Code leicht umschreiben, indem man bestimmte Sequenzen hinzufügt oder herausnimmt.
CRISPRi-Screen
CRISPRi-Screen steht für CRISPR interference screen, ein hochsensibles Werkzeug, durch das Wissenschaftler reduzierte Genexpression untersuchen können.
Deep Sequencing
Deep sequencing ist eine Hochdurchsatz-Sequenzierungstechnik, bei der ein bestimmter genomischer Abschnitt mehrfach analysiert wird. Dies kann hundert- oder tausendmal sein. Diese Technik ermöglicht es Wissenschaftlern, nach Zellen, Mikroben oder seltenen Klontypen zu suchen, die weniger als 1% einer biologischen Probe ausmachen. Dadurch kann sie von Forschern verwendet werden, um in einer Wirtsprobe vorhandene Krankheitserreger zu identifizieren.
Differentielle RNA-Seq
Differentielle RNA-Seq ist eine Hochdurchsatz-Screening-Technik, mit der untersucht wird, ob ein mRNA-Molekül ein primäres oder ein prozessiertes Transkript ist.
Dual RNA-Seq
Dual RNA-Seq ist eine Technik, die die gleichzeitige Analyse von Genen aus zwei verschiedenen Zellen ermöglicht, zum Beispiel die eines Wirts und eines Krankheitserregers. Diese Technik kann zur Untersuchung des gesamten Infektionsprozesses und der Interaktionen zwischen den beiden Zellen eingesetzt werden.
Gen-Fitness
Fitness im genetischen Kontext ist die Quantifizierung des Beitrags eines Genprodukts zum Überleben unter einer bestimmten Bedingung.
Genregulation
Zellen regulieren die Expression ihrer Gene über viele verschiedene Mechanismen, um die Herstellung bestimmter Genprodukte (Proteine oder RNA) zu erhöhen oder zu verringern.
Genetischer Locus
Ein Genetischer Locus ist eine feststehende Stelle auf einem Chromosom, wo sich ein bestimmtes Gen oder eine bestimmte DNA-Sequenz befindet.
Genom-Editierung
Genom-Editierung ist eine Genomtechnik, bei der bestimmte DNA-Segmente einer Zelle eingefügt, entfernt oder modifiziert werden.
Grad-Seq
Der Begriff Grad-Seq bezieht sich auf eine Technik namens RNA gradient profiling by sequencing. Diese Technik ermöglicht es Wissenschaftlern, die codierenden und nichtcodierenden Segmente eines RNA-Transkripts zu analysieren. Grad-Seq ermöglicht die Untersuchung bestimmter funktioneller RNA-Klassen sowie wichtiger RNA-bindender Proteine und ihrer Interaktionen.
Genomweite Chemische Hochdurchsatz-Sondierungstechnologien
Genomweite Chemische Hochdurchsatz-Sondierungstechnologien, die als Werkzeuge und Methoden zur Untersuchung der zahlreichen RNA-Strukturen und -Funktionen auf einem gesamten Genom eingesetzt werden.
In-Vitro-Modelle
In-Vitro-Modelle sind Experimente, die an Mikroorganismen, Zellen oder biologischen Molekülen außerhalb ihres normalen biologischen Kontextes durchgeführt werden. Stattdessen erfolgt das Experiment in der Regel in Reagenzgläsern, Petrischalen, Kolben oder Mikrotiterplatten.
Library Screening
Library Screening bezeichnet die Testung großer Genbibliotheken, die Forschern mit nur einem Experiment gewaltige Datenmengen zur Verfügung stellen. Wissenschaftler nutzen diese Bibliotheks-Screenings, um schnell mehr über die Zielauswahl bei CRISPR-Cas-Systemen zu lernen und um die Genetik verschiedener Organismen über das gesamte Genom hinweg zu untersuchen.
LncRNA
Lange nichtcodierende RNA ist eine Klasse regulatorischer RNA, die Chromosomenstruktur, Transkription, Splicing, die Stabilität der Boten-RNA und ihre Verfügbarkeit beeinflussen kann. LncRNAs können auch zu posttranskriptionellen Veränderungen beitragen.
Maschinelles Lernen
Maschinelles Lernen ist ein Zweig der künstlichen Intelligenz, der Anwendungen entwickelt, die selbstständig aus Daten lernen und ihre Mustererkennung und Vorhersagegenauigkeit im Laufe der Zeit verbessern können. Algorithmen, eine Abfolge von statistischen Programmierschritten, werden dazu "trainiert", Muster in großen Datensätzen zu finden, um Entscheidungen und Vorhersagen auf der Grundlage neuer Daten zu treffen. In der Wissenschaft können Deep-Learning-Algorithmen Merkmale aus sehr großen annotierten Datensätzen, wie beispielsweise einer Sammlung von Genomen, entnehmen, um aus diesen Informationen neue Erkenntnisse zu gewinnen und Vorhersagewerkzeuge auf der Grundlage von Mustern in den Daten zu erstellen.
Massenspektrometrie
Die Massenspektrometrie ist eine Methode, um die Masse geladener Atome oder Moleküle zu bestimmen. Die Teilchen werden dafür ionisiert und anschließend (z.B. durch Magnete) nach ihrem Verhältnis von Masse zu Ladung getrennt. Diese Methode wird benutzt, um Biomoleküle zu untersuchen und chemische Stoffe oder Strukturen zu identifizieren. Da sich die so gewonnenen Daten leicht digitalisieren lassen, werden Massenspektrometer zunehmend direkt mit Computern gekoppelt.
Mikrobiota
Der Begriff Mikrobiota bezieht sich auf eine spezifische Gruppe von Mikroorganismen (Archaeen, Bakterien, Viren und Pilze), die eine bestimmte Umgebung besiedeln. Der menschliche Körper ist dicht mit Mikroorganismen besiedelt, insbesondere die Atemwege, der Urogenital- und der Magen-Darm-Trakt. Die menschliche Mikrobiota kann durch eine Reihe von Faktoren, wie etwa die Ernährung oder den Einsatz von Antibiotika, beeinflusst werden. Das empfindliche Gleichgewicht dieser Mikroorganismenpopulation kann die Gesundheit des Menschen erheblich beeinflussen und dabei sowohl schützende als auch schädliche Auswirkungen haben.
Mikroumgebung
Als Mikroumgebung wird das unmittelbare Umfeld einer Zelle oder eines Gewebes bezeichnet. Sie setzt sich aus der extrazellulären Matrix, Zellen und interstitieller Flüssigkeit zusammen. Die Mikroumgebung beeinflusst den zellulären Phänotypen durch physikalische, mechanische und biochemische Mechanismen.
Mutationsbasierte funktionelle Screens
Mutationsbasierte funktionelle Screens, auch bekannt als Mutations-Interferenz-Mapping-Experimente (MIME), werden verwendet, um mit Einzelnukleotid-Auflösung die Primärsequenz und Sekundärstrukturen eines RNA-Moleküls zu identifizieren, die für dessen Funktion entscheidend sind. MIME basiert auf zufälliger Mutagenese der Ziel-RNA, gefolgt von funktioneller Selektion und Next-Generation-Sequenzierung.
Next-Generation-Sequenzierung
Next-Generation-Sequenzierung oder auch Hochdurchsatzsequenzierung bezeichnet verschiedene moderne Sequenzierungstechnologien. Diese Technologien identifizieren die Nukleotidreihenfolge von DNA oder RNA-Transkripten und quantifizieren sie. Sie ermöglichen eine viel billigere und schnellere Sequenzierung als frühere Techniken und haben das Forschungsgebiet der Genomik und die Molekularbiologie revolutioniert.
Nichtcodierende RNA/Regulatorische RNA
Nichtcodierende RNA bezeichnet eine Gruppe von RNA-Molekülen, die von nichtcodierenden Genen stammen. Sie codieren keine Proteine, besitzen aber andere spezialisierte zelluläre und molekulare Funktionen. Das bedeutet, dass sie nicht wie die Boten-RNA translatiert werden, sondern als RNA-Moleküle innerhalb der Zelle aktiv bleiben. Nichtcodierende RNA-Moleküle spielen eine wichtige Rolle bei der Regulation der Genexpression (An- und Ausschalten) und werden daher auch als regulatorische RNAs bezeichnet. Sie sind an der Regulation der Transkription und Translation von Genen, an posttranskriptionellen Veränderungen und an epigenetischer Regulation beteiligt.
Organoid
Organoide sind organähnliche Mikrostrukturen, die sich in einem Nährmedium aus Stammzellen kultivieren lassen und sich von selbst in unterschiedliche, spezialisierte Gewebe organisieren. Auf diese Weise konnten bereits Modelle für Gehirn, Herz, Niere, Magen und Darm erzeugt werden. Obwohl Organoide in Größe und Komplexität nicht ganzen Organen entsprechen, bieten sie eine exzellente Möglichkeit zur Erforschung von Krankheiten und deren Behandlung.
Pseudoknoten
Ein Pseudoknoten ist eine RNA-Struktur, die durch Verdrehungen und Faltungen des RNA-Strangs aufgrund chemischer Wechselwirkungen innerhalb des Moleküls entsteht. Diese Wechselwirkungen können zu sehr komplexen und stabilen RNA-Strukturen führen. Durch Variationen in der räumlichen Anordnung und den Wechselwirkungen der Schleifen und Faltungen besteht zwischen verschiedenen Pseudoknoten eine große strukturelle Vielfalt. Pseudoknoten haben eine Vielzahl von Funktionen und spielen eine zentrale Rolle bei Veränderungen der Genexpression, indem sie bei vielen Viren einen ribosomalen Frameshift induzieren.
Ribosomales Frameshifting
Ribosomales Frameshifting ist ein Prozess, bei dem die Translation "neu codiert" wird. Das bedeutet, dass der Code in einer Boten-RNA auf alternative Weise gelesen werden kann. Dieses Phänomen tritt nicht durch eine tatsächliche Änderung der Codesequenz auf, sondern einfach dadurch, dass der Lesevorgang (das Raster, englisch frame) von einem anderen Ausgangspunkt aus eingeleitet wird. Da die Nukleotide der Boten-RNA in Dreiergruppen (Codons) gelesen werden, verändert sich durch die Änderung des Leserasters die Art und Weise, wie die gesamte Sequenz decodiert wird. Daraus ergibt sich die Möglichkeit, mehr als ein Protein aus einer einzigen Boten-RNA herzustellen. Dieser Mechanismus wird häufig von Viren genutzt.
Ribosom
Ribosomen sind Zellorganellen, die als Proteinfabriken dienen. Hier findet die Translation der Boten-RNA in Proteine statt. Während der Translation können mehrere Ribosomen an einen einzigen Boten-RNA-Strang gebunden werden. Eine Säugetierzelle kann bis zu 10 Millionen Ribosomen besitzen.
Ribo-Seq
Ribo-Seq ist eine auf RNA-Sequenzierung aufbauende Methode, mit deren Hilfe Wissenschaftler eine „Momentaufnahme“ aller Ribosomen erhalten können, die zu einem bestimmten Zeitpunkt aktiv an der Translation in einer Zelle arbeiten. Dadurch können die Startpunkte der Translation auf mRNA-Molekülen erkannt sowie Einblicke in die Synthese von Peptiden und Proteinen gewonnen werden.
RNA
RNA (Ribonukleinsäure) ist eine Verbindung, die an der Genexpression und der zellulären Proteinsynthese beteiligt ist. Bei bestimmten Viren kann sie auch die DNA (Desoxyribonukleinsäure) als Träger der genetischen Information ersetzen. Wie die DNA besteht sie aus verschiedenen sich wiederholenden Einheiten, den sogenannten Nukleotiden, die die Information codieren. Die Einheiten, aus denen sich die RNA zusammensetzt, sind: Adenosin, Cytosin, Guanin und Uracil.
mRNA
Die Boten-RNA (mRNA, englisch messenger RNA) trägt die codierten Anweisungen, mit denen während der Translation Proteine gebildet werden. Sie wird daher als codierende RNA klassifiziert. Andere RNA-Typen besitzen strukturelle, katalytische und regulatorische Funktionen; diese werden als nichtcodierende RNA klassifiziert. Beispiele für nichtcodierende RNA sind Transfer-RNA (tRNA), ribosomale RNA, kleine RNA und lange nichtcodierende RNA.
RNA-Antisense-Reinigung
Die RNA-Antisense-Reinigung ist eine biochemische Reinigungsmethode, mit der man DNA, RNA oder Proteine identifizieren kann, die mit einer RNA von Interesse assoziiert sind.
RNA-bindendes Protein
RNA-bindende Proteine spielen eine entscheidende Rolle bei der Regulation sowohl der transkriptionellen als auch der posttranskriptionellen Genexpression. Sie können nicht nur den Splicing-Prozess, sondern auch den Transport, die Translation und den Zerfall von Boten-RNA regulieren. RNA-bindende Moleküle sind für die Stabilisierung und Destabilisierung von Boten-RNA als Reaktion auf ihre Umwelt von wesentlicher Bedeutung.
Metabolische RNA-Markierung
Die metabolische RNA-Markierung ist eine Technik, die wertvolle Einblicke in die Geschichte einer Zelle liefert. Die Methode basiert auf dem Einbau von modifizierten Ribonukleosiden in naszierende RNA-Transkripte während der Zellproliferation.
Einzelzell-RNA-Seq
Die Einzelzell-RNA-Sequenzierung ist eine Analysetechnik der Genomik, die für den Nachweis und die Quantifizierung von RNA-Molekülen in einer einzelnen Zelle verwendet wird. Sie ermöglicht es Wissenschaftlern, eine spezifische Zelle zu untersuchen und regulatorische Beziehungen zwischen Genen zu beobachten und bietet einen wertvollen Einblick in die Zellabstammung. Dieser Ansatz ist wichtig, um Zellantworten detailliert und individualisiert zu analysieren und zu verstehen.
RNA-Prozessierung
Bevor bei Eukaryoten die mRNA den Zellkern für die Proteinbiosynthese verlässt, wird sie prozessiert. Sie erhält am einen Ende eine „Kappe“ (5′-Cap-Struktur) und am anderen Ende einen Poly-A-Schwanz aus Adenin-Nukleotiden. Beide Strukturen schützen die mRNA und helfen bei der Translation. Hinzu kommt der Splicing-Prozess, bei dem die nicht mehr relevanten Introns aus der mRNA entfernt und die verbleibenden Exons miteinander verbunden werden.
RNA-Seq
RNA-Seq ist die Bestimmung der Menge und Nukleotidabfolge der RNA in einer Probe durch Next-Generation-Sequencing. Dadurch können beispielsweise Informationen zur Genexpression und zu posttranskriptionalen Modifikationen erlangt werden. Neue Methoden der RNA-Sequenzierung sind die Einzelzellsequenzierung und Dual RNA-seq.
Kleine RNA
Kleine RNAs sind eine Klasse regulatorischer RNAs, die eine entscheidende Rolle bei der posttranskriptionellen Regulation der Genexpression spielen. Sie können die Stabilität und Regulation der Boten-RNA kontrollieren und zu epigenetischen Veränderungen beitragen.
Splicing
Splicing ist der Prozess, bei dem die nichtcodierenden Abschnitte der Gene, die sogenannten Introns, aus der ursprünglichen Boten-RNA entfernt werden. Zurück bleiben nur die codierenden Segmente, Exons genannt, die dann wieder miteinander verbunden werden. Durch das Splicing entsteht eine reife Boten-RNA, die für die Proteinsynthese bereit ist.
Synthetische Biologie
Synthetische Biologie ist ein multidisziplinäres Forschungsgebiet, in dem biologische Teile oder Systeme für nützliche Zwecke entworfen oder angepasst werden, damit sie neue Eigenschaften haben. Dieses Ziel kann erreicht werden, indem man neue Systeme erschafft oder bereits existierende Systeme umgestaltet.
Transkriptom
Als Transkriptom werden alle von DNA in RNA umgeschriebenen Gene in einer Zelle bezeichnet. Dies kann sowohl kodierende als auch nichtkodierende RNA sein. In der Transkriptomik wird das Transkriptom untersucht, um beispielsweise die Genexpression genauer zu verstehen.
Transkriptomik
Transkriptomik ist die Untersuchung aller RNA-Moleküle innerhalb einer Zelle, die auch als das Transkriptom bezeichnet werden. Während sich viele Studien in der Transkriptomik auf Boten-RNA konzentrieren, die für Proteine codiert, ist ein großer Teil der RNAs in einer Zelle nichtcodierend und hat stattdessen eine regulierende Rolle. Räumliche Transkriptomik ermöglicht es Wissenschaftlern, Genaktivitäten in einer Zell- oder Gewebeprobe zu erkennen und zu verorten.
Translation
Translation ist der Prozess, durch den aus einer Boten-RNA-Vorlage Proteine gebildet werden. Dies geschieht innerhalb des Ribosoms, wo die Nukleotidsequenz von der Vorlage abgelesen wird. Das Lesen ist ein streng kontrollierter Prozess: Die Nukleotide werden in Tripletts, sogenannten Codons, in einer bestimmten Richtung abgelesen. Jedes Codon wird in seine entsprechende Aminosäure übersetzt. Auf diese Weise wird eine Kette von Aminosäuren in einer bestimmten, von der Boten-RNA vorgegebenen Reihenfolge synthetisiert, um ein Protein zu formen.
Transposon-Mutagenese
Die Transposon-Mutagenese ist eine Methode, um zufällige Gene zu inaktivieren. Dafür wird ein eigennütziges DNA-Element verwendet, das sich zu unterschiedlichen Orten im Genom bewegen kann.
Virionen
Virionen sind Viruspartikel, die sich außerhalb der Zelle befinden. Sie enthalten Nukleinsäuren (DNA oder RNA) und werden meist von einer Proteinkapsel sowie manchmal von einer Biomembran umschlossen. Mangels eines eigenen Stoffwechsels müssen Virionen in Zellen eindringen, um sich (dann als Virus) vermehren zu können.
Virulenz
Virulenz beschreibt, wie aggressiv, schädigend und krankmachend ein Erreger ist. Die Virulenz hängt von vielen verschiedenen Faktoren ab. Sie wird durch Virulenzfaktoren des Erregers bestimmt, die ihm helfen, sich im infizierten Wirt auszubreiten.