Prof. Neva Caliskan, Ehemalige Gruppenleiterin
Rekodierungsmechanismen in Infektionen
Im Jahr 2018 richtete das Helmholtz-Institut Würzburg (HIRI) eine Forschungsgruppe für Rekodierungsmechanismen in Infektionen unter der Leitung von Neva Caliskan ein. Caliskan, die 2024 zur Professorin und Leiterin des Lehrstuhls für Biochemie III an der Universität Regensburg ernannt wurde, unterhält weiterhin enge Verbindungen zur Forschung am HIRI.
Unsere Forschung
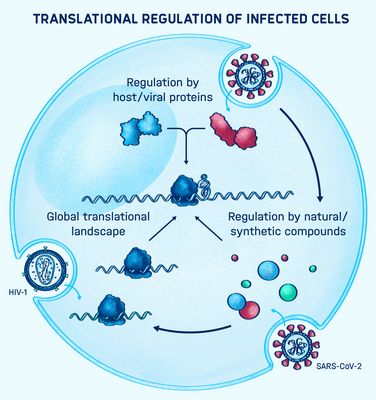
In Viren oder zellulären Genen verschlüsselte RNAs können während der Translation auf alternative Weise gelesen werden, was als Rekodierung bezeichnet wird. Es ist jedoch unklar, wie genau die Rekodierung durch den Wirt reguliert wird. Ein genaues Verständnis der Rekodierung und ihrer Regulation kann daher der Schlüssel zur Entwicklung neuer RNA-basierter Therapien im Kampf gegen Infektionen sein.
In diesem Zusammenhang untersuchte Neva Caliskans Gruppe die Funktion und Dynamik von RNA-Molekülen und wie sie mit Faktoren mit Trans-Wirkung zusammenspielen. Die Wissenschaftler arbeiteten dafür mit Viren wie Corona- und Retroviren, bei denen bekannt ist, dass sie für ihre Replikation auf Rekodierung angewiesen sind. Gemeinsam entwickelten sie Methoden, um RNA-Komplexe und Translation in bisher ungekannter Detailtreue zu erforschen.
Die Gruppe nutzte ein stark interdisziplinäres Toolset, etwa RNA-Antisense-Reinigung und Massenspektrometrie, um RNA-Interaktionspartner zu erkennen sowie zelluläre Assays um molekulare Details zu untersuchen. Assays für Einzelmoleküle oder Molekülgruppen wie optische Pinzetten spielen für die Erforschung von RNA-Komplexen eine wichtige Rolle. Um die Entwicklung RNA-basierter Therapien voranzubringen, wollten die Wissenschaftlerinnen und Wissenschaftler verstehen, wie RNA-Strukturelemente mit anderen Faktoren in der Zelle zusammenwirken, um die Weise zu regulieren, wie mRNA von den Ribosomen gelesen wird.
Team-Mitglieder

Prof. Neva Caliskan, Ehemalige Gruppenleiterin
Gruppenleiterin
Forschungsprojekte
Alumni
Stefan Buck, Data Manager • Orian Gilmer, Postdoc • Anuja Kibe, Doktorandin • Tatyana Koch, Technische Assistentin • Saurav Malik, Doktorand • Rui Pedro Marques da Costa, Postdoc • Lukáš Pekárek, Doktorand • Ricarda Riegger, Doktorandin • Alexandria Smart, Postdoc • Matthias Zimmer, Doktorand & Postdoc
Publikationen
2025
Guidelines for minimal reporting requirements, design and interpretation of experiments involving the use of eukaryotic dual gene expression reporters (MINDR)
Loughran G, Andreev DE, Terenin IM, Namy O, Mikl M, Yordanova MM, McManus CJ, Firth AE, Atkins JF, Fraser CS, …, Dmitriev SE, Baranov PV (2025)
Nature Structural & Molecular Biology 32 (3): 418-430
Optimization of Structure-Guided Development of Chemical Probes for the Pseudoknot RNA of the Frameshift Element in SARS-CoV-2
Ceylan B, Adam J, Toews S, Kaiser F, Dörr J, Scheppa D, Tants JN, Smart A, Schoth J, Philipp S, …, Wacker A, Schwalbe H (2025)
Angewandte Chemie International Edition 64 (9): e202417961
AcrVIB1 inhibits CRISPR-Cas13b immunity by promoting unproductive crRNA binding accessible to RNase attack
Wandera KG, Schmelz S, Migur A, Kibe A, Lukat P, Achmedov T, Caliskan N, Blankenfeldt W, Beisel CL (2025)
Molecular Cell 85 (6): 1162-1175
The translational landscape of HIV-1 infected cells reveals key gene regulatory principles
Kibe A, Buck S, Gribling-Burrer AS, Gilmer O, Bohn P, Koch T, Mireisz CN, Schlosser A, Erhard F, Smyth RP, Caliskan N (2025)
Nature Structural & Molecular Biology 32 (5): 841-852
Designing the Aplysia punctata Arginine-Depleting Enzyme for Tumor Targeting
Wolkersdorfer AM, Endo Y, Kehrein J, Kappus M, Hattori S, Gutmann M, Rudel T, Caliskan N, Lühmann T, Kato Y, Meinel L (2025)
Molecular Pharmaceutics 22 (3): 1253–1261
Giant RNA genomes: Roles of host, translation elongation, genome architecture, and proteome in nidoviruses
Neuman BW, Smart A, Gilmer O, Smyth RP, Vaas J, Böker N, Samborskiy DV, Bartenschlager R, Seitz S, Gorbalenya AE, Caliskan N, Lauber C (2025)
Proceedings of the National Academy of Sciences of the United States of America 122 (7): e2413675122
2024
Translation Inhibition Mediated by Interferon-Stimulated Genes during Viral Infections
Smart A, Gilmer O, Caliskan N (2024)
Viruses 16 (7)
2023
Cis-mediated interactions of the SARS-CoV-2 frameshift RNA alter its conformations and affect function
Pekarek L, Zimmer MM, Gribling-Burrer AS, Buck S, Smyth RP, Caliskan N (2023)
Nucleic Acids Research 51 (2): 728–743
SND1 binds SARS-CoV-2 negative-sense RNA and promotes viral RNA synthesis through NSP9
Schmidt N, Ganskih S, Wei Y, Gabel A, Zielinski S, Keshishian H, Lareau CA, Zimmermann L, Makroczyova J, Pearce C, …, Erhard F, Munschauer M (2023)
Cell 186 (22): 4834-4850.e23
Mouse Liver-Expressed Shiftless Is an Evolutionarily Conserved Antiviral Effector Restricting Human and Murine Hepaciviruses
Zhang Y, Kinast V, Sheldon J, Frericks N, Todt D, Zimmer M, Caliskan N, Brown RJP, Steinmann E, Pietschmann T (2023)
Microbiology Spectrum 11 (4): e0128423
2022
Short- and long-range interactions in the HIV-1 5' UTR regulate genome dimerization and packaging
Ye L, Gribling-Burrer AS, Bohn P, Kibe A, Börtlein C, Ambi UB, Ahmad S, Olguin-Nava M, Smith M, Caliskan N, von Kleist M, Smyth RP (2022)
Nature Structural & Molecular Biology 29 (4): 306-319
Spacer prioritization in CRISPR-Cas9 immunity is enabled by the leader RNA
Liao C, Sharma S, Svensson SL, Kibe A, Weinberg Z, Alkhnbashi OS, Bischler T, Backofen R, Caliskan N, Sharma CM, Beisel CL (2022)
Nature Microbiology 7 (4): 530-541
Editorial: mRNA Translational Control as a Mechanism of Post-transcriptional Gene Regulation
Kiss DL, Vasudevan D, Ho CK, Caliskan N (2022)
Frontiers in Molecular Biosciences 9: 947516
POTATO: Automated pipeline for batch analysis of optical tweezers data
Buck S, Pekarek L, Caliskan N (2022)
Biophysical Journal 121 (15): 2830-2839
Insights from structural studies of the cardiovirus 2A protein
Caliskan N, Hill CH (2022)
Bioscience Reports 42 (1): BSR20210406
Optical Tweezers to Study RNA-Protein Interactions in Translation Regulation
Pekarek L, Buck S, Caliskan N (2022)
Journal of Visualized Experiments (180)
Thinking Outside the Frame: Impacting Genomes Capacity by Programmed Ribosomal Frameshifting
Riegger RJ, Caliskan N (2022)
Frontiers in Molecular Biosciences 9: 842261
2021
Structural and molecular basis for Cardiovirus 2A protein as a viral gene expression switch
Hill CH, Pekarek L, Napthine S, Kibe A, Firth AE, Graham SC, Caliskan N, Brierley I (2021)
Nature Communications 12 (1): 7166
Investigating molecular mechanisms of 2A-stimulated ribosomal pausing and frameshifting in Theilovirus
Hill CH, Cook GM, Napthine S, Kibe A, Brown K, Caliskan N, Firth AE, Graham SC, Brierley I (2021)
Nucleic Acids Research 49 (20): 11938-11958
The short isoform of the host antiviral protein ZAP acts as an inhibitor of SARS-CoV-2 programmed ribosomal frameshifting
Zimmer MM, Kibe A, Rand U, Pekarek L, Ye L, Buck S, Smyth RP, Cicin-Sain L, Caliskan N (2021)
Nature Communications 12 (1): 7193
2020
The SARS-CoV-2 RNA-protein interactome in infected human cells
Schmidt N, Lareau CA, Keshishian H, Ganskih S, Schneider C, Hennig T, Melanson R, Werner S, Wei Y, Zimmer M, …, Bodem J, Munschauer M (2020)
Nature Microbiology 6 (3): 339-353
2019
Thermodynamic control of -1 programmed ribosomal frameshifting
Bock LV, Caliskan N, Korniy N, Peske F, Rodnina MV, Grubmüller H (2019)
Nature Communications 10: 4598
2018
Small synthetic molecule-stabilized RNA pseudoknot as an activator for -1 ribosomal frameshifting
Matsumoto S, Caliskan N, Rodnina MV, Murata A, Nakatani K (2018)
Nucleic Acids Research 46 (16): 8079-8089
2017
Conditional Switch between Frameshifting Regimes upon Translation of dnaX mRNA
Caliskan N, Wohlgemuth I, Korniy N, Pearson M, Peske F, Rodnina MV (2017)
Molecular Cell 66 (4): 558-567.e4
2016
Choreography of molecular movements during ribosome progression along mRNA
Belardinelli R, Sharma H, Caliskan N, Cunha CE, Peske F, Wintermeyer W, Rodnina MV (2016)
Nature Structural & Molecular Biology 23 (4): 342-8
2015
Changed in translation: mRNA recoding by -1 programmed ribosomal frameshifting
Caliskan N, Peske F, Rodnina MV (2015)
Trends in Biochemical Sciences 40 (5): 265-74
2014
Programmed -1 frameshifting by kinetic partitioning during impeded translocation
Caliskan N, Katunin VI, Belardinelli R, Peske F, Rodnina MV (2014)
Cell 157 (7): 1619-31