Prof. Franziska Faber
RNA-Biologie gram-positiver Bakterien (assoziierte Forschungsgruppe)
Unsere Forschung
Franziska Faber leitet eine Nachwuchsgruppe der Julius-Maximilians-Universität Würzburg, die mit dem HIRI assoziiert ist. Das Team erforscht das Zusammenspiel von Clostridioides difficile, einem Darmpathogen, und der intestinalen Mikrobiota. Ziel ist es, die RNA-basierten Kontrollmechanismen der Virulenz während dieser Wechselwirkungen besser zu verstehen. Dieses Wissen soll dann für die Entwicklung neuer antimikrobieller Wirkstoffe auf RNA-Basis genutzt werden.
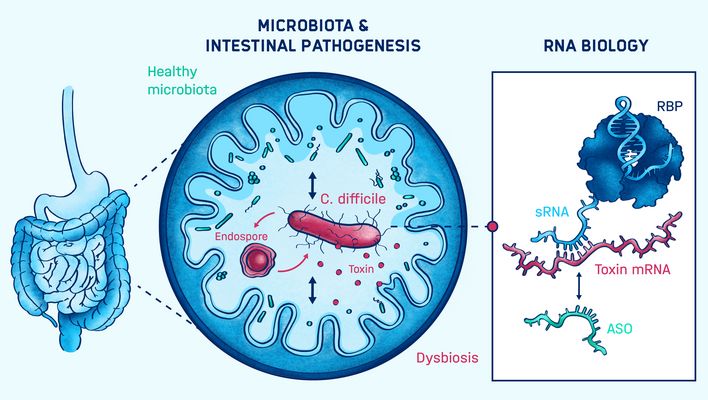
Darminfektionen stehen häufig in Zusammenhang mit einer zugrundeliegenden mikrobiellen Dysbiose, also einem Ungleichgewicht der Darmflora. Ein gutes Beispiel ist das Bakterium Clostridioides difficile – eine der Hauptursachen für Antibiotika-assoziierte Durchfallerkrankungen: Bei gesunden Menschen verläuft die Infektion mit C. difficile symptomlos. Nach einer Antibiotikabehandlung verursacht das Darmpathogen jedoch ein breites Spektrum an Krankheiten. Daher ist ein grundlegendes Verständnis der Virulenzregulation im Kontext von Pathogen-Mikrobiota-Interaktionen von entscheidender Bedeutung für die Entwicklung neuer RNA-basierter Therapeutika.
Die Gruppe um Franziska Faber verfolgt dieses Ziel durch die molekulare Charakterisierung der Virulenzregulation, wobei ihr Schwerpunkt auf der posttranskriptionellen Regulation durch nicht-kodierende RNAs und deren RNA-bindenden Proteinen liegt. Außerdem charakterisiert sie mikrobielle und Wirtssignale, die die Expression von Virulenzgenen regulieren. Weiter erforscht die Faber-Gruppe, wie Gene und Signalwege, die für die Virulenz wichtig sind, mithilfe von Antisense-Oligomeren modifiziert werden können.
Bei seiner Arbeit kombiniert das Team Technologien aus den Bereichen der RNA-Biologie, Biochemie, Metabolomik, Genetik und Mikrobiologie. Das übergeordnete Ziel der Faber-Gruppe ist die Identifizierung und Charakterisierung RNA-basierter Mechanismen der Virulenzregulation, die als therapeutische Angriffspunkte genutzt werden können.
Team-Mitglieder

Prof. Franziska Faber
Gruppenleiterin

Christopher Birk
Doktorand

Jakob Jeschonneck
Doktorand

Janet Wackenreuter
Doktorandin

Johannes Sulzer
Doktorand

Jonathan Scheler
Doktorand

Sara Giddins
Doktorandin

Anna Zweyer
Technische Assistentin
Forschungsprojekte
Die Bildung von Toxinen und die Fähigkeit, antibiotikaresistente Sporen zu erzeugen, sind die wichtigsten Virulenzfaktoren von C. difficile. Diese tragen zu erheblicher Morbidität, Mortalität sowie zu hohen Rezidivraten bei Patient:innen bei. Ihre Produktion wird durch Stoffwechselsignale gesteuert, deren Abundanz und Zusammensetzung während des Infektionszyklus von der Darmmikrobiota beeinflusst wird.
In unserem Labor kombinieren wir auf RNA-Sequenzierung basierende Ansätze mit RNA-Biologie, biochemischen, genetischen und mikrobiologischen Techniken. Unser Ziel ist es, nicht-kodierende RNAs und deren RNA-bindende Proteinpartner, die an der Regulation von Virulenzgenen beteiligt sind, zu identifizieren und zu charakterisieren.
Ferner sind wir daran interessiert, wie die Aktivitäten dieser Regulatoren durch mikrobielle und Wirtssignale beeinflusst werden. Um diese Wechselwirkungen besser zu verstehen, verfolgen wir einen Bottom-up-Ansatz, bei dem wir einzelne Interaktionen zwischen Erreger und Wirt untersuchen. Dazu kombinieren wir mikrobiologische, genetische, Zellkultur- und Metabolomik-Methoden.
Das übergeordnete Ziel unserer Forschung ist es, regulatorische RNAs und ihre Signalwege zu identifizieren, um diese in Zukunft als therapeutische Angriffspunkte nutzen zu können. Um dieses Ziel zu erreichen, entwickeln wir derzeit eine auf Antisense-Oligomeren basierende Gen-Abschaltung in C. difficile. Dabei konzentrieren wir uns auf essenzielle Gene, also Erbgutabschnitte, ohne die der Erreger nicht leben kann.
Alumni
Vanessa Lamm-Schmidt, Doktorandin • Tina Lence, Postdoc
Publikationen
2025
Enabling next-generation anaerobic cultivation through biotechnology to advance functional microbiome research
Clavel T, Faber F, Groussin M, Haller D, Overmann J, Pauvert C, Poyet M, Selkrig J, Stecher B, Typas A, …, Wylensek D, Maier L (2025)
Nature Biotechnology (Online ahead of print)
The global RNA-binding protein RbpB is a regulator of polysaccharide utilization in Bacteroides thetaiotaomicron
Rüttiger AS, Ryan D, Spiga L, Lamm-Schmidt V, Prezza G, Reichardt S, Langford M, Barquist L, Faber F, Zhu W, Westermann AJ (2025)
Nature Communications 16 (1): 208
An sRNA overexpression library reveals AbnZ as a negative regulator of an essential translocation module in Caulobacter crescentus
Velasco-Gomariz M, Sulzer J, Faber F, Fröhlich KS (2025)
Nucleic Acids Research 53 (1): gkae1139
2024
Formation of the pyruvoyl-dependent proline reductase Prd from Clostridioides difficile requires the maturation enzyme PrdH
Behlendorf C, Diwo M, Neumann-Schaal M, Fuchs M, Körner D, Jänsch L, Faber F, Blankenfeldt W (2024)
PNAS nexus 3 (7): pgae249
The conserved noncoding RNA ModT coordinates growth and virulence in Clostridioides difficile
Lence T, Sulzer J, Andress K, Gribling-Burrer AS, Lamm-Schmidt V, Barquist L, Smyth RP, Faber F (2024)
PLOS Biology 22 (12): e3002948
2023
Durchfallerreger und die Homöostase der Mikrobiota
Winter S, Faber F, Bäumler A (2023)
BIOspektrum 29 (2): 127-129
A network of small RNAs regulates sporulation initiation in C difficile
Fuchs M, Lamm-Schmidt V, Lence T, Sulzer J, Bublitz A, Wackenreuter J, Gerovac M, Strowig T, Faber F (2023)
EMBO Journal 42 (12): e112858
2021
RNA landscape of the emerging cancer-associated microbe Fusobacterium nucleatum
Ponath F, Tawk C, Zhu Y, Barquist L, Faber F, Vogel J (2021)
Nature Microbiology 6 (8): 1007-1020
Grad-seq identifies KhpB as a global RNA-binding protein in Clostridioides difficile that regulates toxin production
Lamm-Schmidt V, Fuchs M, Sulzer J, Gerovac M, Hör J, Dersch P, Vogel J, Faber F (2021)
microLife 2: uqab004
Malaria parasite infection compromises colonization resistance to an enteric pathogen by reducing gastric acidity
Walker GT, Yang G, Tsai JY, Rodriguez JL, English BC, Faber F, Souvannaseng L, Butler BP, Tsolis RM (2021)
Science Advances 7 (27): eabd6232
The metabolic footprint of Clostridia and Erysipelotrichia reveals their role in depleting sugar alcohols in the cecum
Tiffany CR, Lee JY, Rogers AWL, Olsan EE, Morales P, Faber F, Bäumler AJ (2021)
Microbiome 9 (1): 174
An RNA-centric global view of Clostridioides difficile reveals broad activity of Hfq in a clinically important gram-positive bacterium
Fuchs M, Lamm-Schmidt V, Sulzer J, Ponath F, Jenniches L, Kirk JA, Fagan RP, Barquist L, Vogel J, Faber F (2021)
PNAS 118 (25): e2103579118
2020
Antibacterial Anacardic Acid Derivatives
Saedtler M, Förtig N, Ohlsen K, Faber F, Masota N, Kowalick K, Holzgrabe U, Meinel L (2020)
ACS Infectious Diseases 6 (7): 1674-1685
2019
Commensal Enterobacteriaceae Protect against Salmonella Colonization through Oxygen Competition
Litvak Y, Mon KKZ, Nguyen H, Chanthavixay G, Liou M, Velazquez EM, Kutter L, Alcantara MA, Byndloss MX, Tiffany CR, …, Zhou H, Bäumler AJ (2019)
Cell Host & Microbe 25 (1): 128-139.e5
Endogenous Enterobacteriaceae underlie variation in susceptibility to Salmonella infection
Velazquez EM, Nguyen H, Heasley KT, Saechao CH, Gil LM, Rogers AWL, Miller BM, Rolston MR, Lopez CA, Litvak Y, …, Byndloss AJ, Bäumler AJ (2019)
Nature Microbiology 4 (6): 1057-1064
Social behaviour and making attachments: a report from the fifth 'Young Microbiologists Symposium on Microbe Signalling, Organisation and Pathogenesis'
An SQ, Ding YC, Faber F, Hobley L, Sá-Pessoa J (2019)
Microbiology 165 (2): 138-145
2018
Genetic Ablation of Butyrate Utilization Attenuates Gastrointestinal Salmonella Disease
Bronner DN, Faber F, Olsan EE, Byndloss MX, Sayed NA, Xu G, Yoo W, Kim D, Ryu S, Lebrilla CB, Bäumler AJ (2018)
Cell Host & Microbe 23 (2): 266-273.e4
2017
Microbiota-activated PPAR-γ signaling inhibits dysbiotic Enterobacteriaceae expansion
Byndloss MX, Olsan EE, Rivera-Chávez F, Tiffany CR, Cevallos SA, Lokken KL, Torres TP, Byndloss AJ, Faber F, Gao Y, …, Lebrilla CB, Bäumler AJ (2017)
Science 357 (6351): 570-575
Colonization resistance: The deconvolution of a complex trait
Olsan EE, Byndloss MX, Faber F, Rivera-Chávez F, Tsolis RM, Bäumler AJ (2017)
The Journal of Biological Chemistry 292 (21): 8577-8581
A Salmonella Regulator Modulates Intestinal Colonization and Use of Phosphonoacetic Acid
Elfenbein JR, Knodler LA, Schaeffer AR, Faber F, Bäumler AJ, Andrews-Polymenis HL (2017)
Frontiers in Cellular and Infection Microbiology 7: 69
Respiration of Microbiota-Derived 1,2-propanediol Drives Salmonella Expansion during Colitis
Faber F, Thiennimitr P, Spiga L, Byndloss MX, Litvak Y, Lawhon S, Andrews-Polymenis HL, Winter SE, Bäumler AJ (2017)
PLOS Pathogens 13 (1): e1006129
2016
Depletion of Butyrate-Producing Clostridia from the Gut Microbiota Drives an Aerobic Luminal Expansion of Salmonella
Rivera-Chávez F, Zhang LF, Faber F, Lopez CA, Byndloss MX, Olsan EE, Xu G, Velazquez EM, Lebrilla CB, Winter SE, Bäumler AJ (2016)
Cell Host & Microbe 19 (4): 443-54
Host-mediated sugar oxidation promotes post-antibiotic pathogen expansion
Faber F, Tran L, Byndloss MX, Lopez CA, Velazquez EM, Kerrinnes T, Nuccio SP, Wangdi T, Fiehn O, Tsolis RM, Bäumler AJ (2016)
Nature 534 (7609): 697-9
2015
Inflammation-associated alterations to the intestinal microbiota reduce colonization resistance against non-typhoidal Salmonella during concurrent malaria parasite infection
Mooney JP, Lokken KL, Byndloss MX, George MD, Velazquez EM, Faber F, Butler BP, Walker GT, Ali MM, Potts R, …, Luckhart S, Tsolis RM (2015)
Scientific Reports 5: 14603
2014
The impact of intestinal inflammation on the nutritional environment of the gut microbiota
Faber F, Bäumler AJ (2014)
Immunology Letters 162 (2 Pt A): 48-53
Salmonella enterica Serovar Typhi conceals the invasion-associated type three secretion system from the innate immune system by gene regulation
Winter SE, Winter MG, Poon V, Keestra AM, Sterzenbach T, Faber F, Costa LF, Cassou F, Costa EA, Alves GE, …, Santos RL, Bäumler AJ (2014)
PLOS Pathogens 10 (7): e1004207
2012
Surface-associated motility, a common trait of clinical isolates of Acinetobacter baumannii, depends on 1,3-diaminopropane
Skiebe E, de Berardinis V, Morczinek P, Kerrinnes T, Faber F, Lepka D, Hammer B, Zimmermann O, Ziesing S, Wichelhaus TA, …, Pfeifer Y, Wilharm G (2012)
International Journal of Medical Microbiology 302 (3): 117-28
2010
A simple and rapid method of bacterial transformation
Wilharm G, Lepka D, Faber F, Hofmann J, Kerrinnes T, Skiebe E (2010)
Journal of Microbiological Methods 80 (2): 215-6
Orbus hercynius gen. nov., sp. nov., isolated from faeces of wild boar, is most closely related to members of the orders 'Enterobacteriales' and Pasteurellales
Volkmann M, Skiebe E, Kerrinnes T, Faber F, Lepka D, Pfeifer Y, Holland G, Bannert N, Wilharm G (2010)
International Journal of Systematic and Evolutionary Microbiology 60 (Pt 11): 2601-2605
2009
CsrA and CsrB are required for the post-transcriptional control of the virulence-associated effector protein AvrA of Salmonella enterica
Kerrinnes T, Zelas ZB, Streckel W, Faber F, Tietze E, Tschäpe H, Yaron S (2009)
International Journal of Medical Microbiology 299 (5): 333-41
2007
Influence of poly(L-lysine) on the structure of dipalmitoylphosphatidylglycerol/water dispersions studied by X-ray scattering
Förster G, Schwieger C, Faber F, Weber T, Blume A (2007)
European Biophysics Journal 36 (4-5): 425-35
2006
Lack of angiotensin II conversion to angiotensin III increases water but not alcohol consumption in aminopeptidase A-deficient mice
Faber F, Gembardt F, Sun X, Mizutani S, Siems WE, Walther T (2006)
Regulatory Peptides 136 (1-3): 130-7