Prof. Antoine-Emmanuel Saliba
Einzelzellanalyse
Unsere Forschung
Die Arbeitsgruppe um Emmanuel Saliba widmet sich der Erforschung von Wirt-Pathogen-Interaktionen in hoher Auflösung auf der Ebene der einzelnen Zelle. Um den Zustand einzelner Erreger zu bestimmen und dadurch die individuellen Unterschiede von Wirtsreaktionen und Krankheitsverläufen zu verstehen, entwickelt und kombiniert das Team Ansätze aus der Einzelzellanalyse mit bildgebenden Verfahren und computergestützten Methoden.
Neueste technologische Fortschritte machen es Wissenschaftlern möglich, die Gesamtheit aller Zellen des Körpers zu kartieren, ihre Zustände zu erfassen und ihre Reaktionen auf infektiöse Erreger in nie gekanntem Detail zu beobachten. Doch was dazu führt, dass ein Wirt die Ausbreitung eines Erregers entweder eindämmt oder aber ein Teil der Erreger bei einer Infektion der Immunüberwachung des Wirts entgehen, ist noch immer weitgehend unverstanden.
Die Arbeitsgruppe von Emmanuel Saliba analysiert RNA und ihre Prozessierung, um den Zustand der Zellen von Wirten und Krankheitserregern auf Einzelzellniveau zu bestimmen. Unter Verwendung von Salmonellen und Atemwegsviren in Kombination mit Zellkulturmodellen, Organoiden und klinischen Proben, analysiert und kategorisiert das Team Zellen, um deren Mikroumgebung zu analysieren und die Heterogenität von Krankheitsverläufen zu verstehen.
Die Wissenschaftler setzen Einzelzell-RNA-seq, räumliche Transkriptomik und RNA-Bildgebungsverfahren ein, um die Gesamtheit an RNA-Transkripten zu erfassen, die in einem Wirt und einem Pathogen während einer Infektion gebildet werden. Einzelzellanalysen im zeitlichen Verlauf und unter Verwendung von metabolischer RNA-Markierung geben weitere Einblicke in die Herkunft und Entwicklung einer Zelle. Durch diese hochauflösenden Analysen ist es möglich, das Verhalten von Zellen vorherzusagen und regulatorische Netzwerke zu entschlüsseln, die Infektionen zugrunde liegen. Diese Arbeit ist entscheidend für die Entwicklung von Präzisionsdiagnostik und -therapeutika.
Team-Mitglieder

Prof. Antoine-Emmanuel Saliba
Gruppenleiter

Alexander Leipold
Doktorand

Anastasiya Grinko
Doktorandin

Ariane Binder
Doktorandin

Chia-Ling Chou
Doktorandin

Christophe Toussaint
Doktorand

Laura Cecchino
Doktorandin

Lisa Chiggiato
Doktorandin

Oliver Dietrich
Doktorand

Tobias Krammer
Doktorand

Tristan Beste
Doktorand

Nina DiFabion
Technische Assistentin
Forschungsprojekte
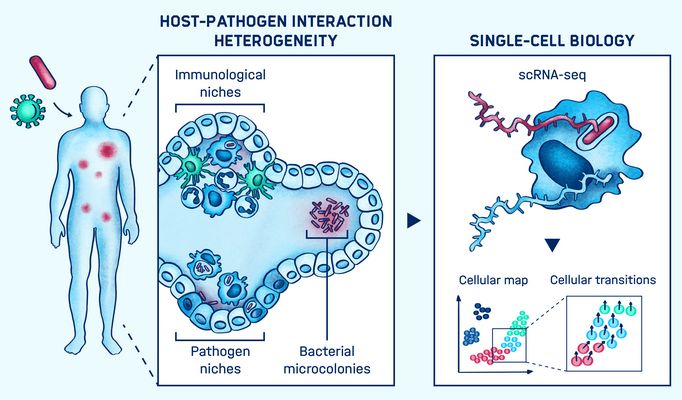
Andauernde bakterielle Infektionen werden durch eine kleine Subpopulation intrazellulärer Erreger verursacht, den „Persistern“ (von Englisch persist: überdauern). Diese können sich über Jahre in verschiedenen Zelltypen und Gewebsorten aufhalten, ohne Symptome zu verursachen. Diese Subpopulationen organisieren sich auf Zellebene in geschützten Nischen und können sich so der Immunüberwachung und medikamentöser Behandlung entziehen. Histologische Studien haben komplexe Gewebsumgestaltungen während Infektionen beschrieben und in neueren in-vivo-Studien auf Einzelzellebene gibt es erste Hinweise auf die Heterogenität der Infektionsherde.
Es bleiben jedoch offene Fragen: Wie sieht die zelluläre Architektur der Infektionsherde genau aus? Wie lassen sich in komplex strukturiertem Gewebe Nischen identifizieren, die sich auf den Krankheitsausgang auswirken? Es gibt erste Annahmen. Etwa, dass Salmonellen in viele Zelltypen eindringen und dort überdauern können. Diese Zelltypen untergliedern sich wiederum in eine Vielzahl verschiedener Unterklassen, deren Existenz und Funktion bis vor kurzem nicht beachtet wurden. Ähnlich verlassen in infizierten Gewebestrukturen wie der Milz viele infizierte Zellen ihre ursprünglichen Entzündungsherde (Läsionen). Von dort aus verbreiten sie sich in verschiedene Gewebe. Um die Heterogenität der infizierten Zellen, deren Mikroumgebung und Funktion zu verstehen, sind Einzelzellstudien in einem In-vivo-Kontext notwendig. Die Gruppe Einzelzellanalyse entwickelt und kombiniert In-vitro- und In-vivo-Einzelzell-Transkriptomik. So arbeitet sie daran, die zelluläre Organisation von Infektionsherden und ihre funktionellen Konsequenzen für die Infektion zu entschlüsseln.
Die noch junge genomweite Einzelzell-Transkriptomik ist eine leistungsfähige Technologie zur Entschlüsselung der Identität und der Funktionen von Zellen. So ermöglicht sie die detaillierte Untersuchung von Heterogenität. Unterstützt wird dieser Forschungszweig durch die Entwicklung automatisierter Plattformen. Diese machen es möglich, tausende Einzelzellen gleichzeitig zu untersuchen. Die Arbeitsgruppe Saliba hat Pionierarbeit beim Einsatz von Einzelzell-RNA-seq im Zusammenhang mit Infektionen geleistet: Sie untersucht die Heterogenität in der Reaktion von Maus-Makrophagen auf Salmonellen. Dabei hat sie sich auf Bakterien mit unterschiedlichem Wachstumsstatus konzentriert, einschließlich nicht wachsender „Persister“, die mit wiederkehrenden Infektionen in Verbindung gebracht wurden. Dabei konnte die Arbeitsgruppe zeigen, wie Salmonellen das breite Spektrum der Wirtspolarisation beeinflussen, und hat die Existenz einer Untergruppe von Makrophagen nachgewiesen, die Entzündungs- und Immunaktivierungsprogrammen entgehen. Diese Studie lieferte neue Einblicke in die Wirtsreaktion, beschränkte sich aber auf die Analyse infizierter Zellen aus in-vitro-Kulturen. Die nächste Herausforderung ist nun die Entschlüsselung der Reaktion einzelner Zellen in infiziertem Gewebe.
Darüber hinaus arbeitet die Gruppe daran, das volle Potenzial der Einzelzell-RNA-Sequenzierung für die infektionsbiologische Grundlagenforschung zu erschließen.
Im Fokus
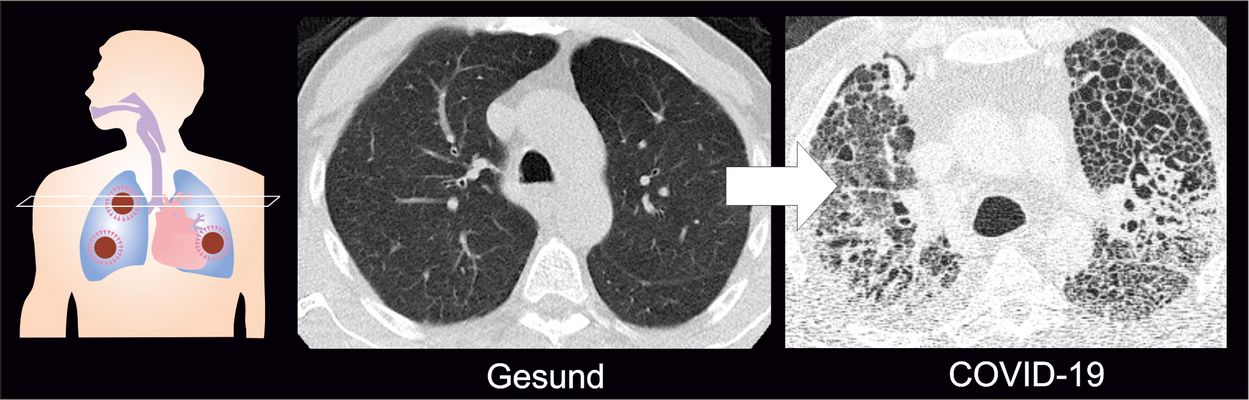
COVID-19-Lungenversagen: Warum Betroffene so lange beatmet werden müssen
Die meisten Patienten mit schwerer COVID-19-Infektion entwickeln eine ungewöhnlich ausgeprägte Narbenbildung in der Lunge. In einer gemeinsamen, in der Fachzeitschrift Cell veröffentlichten Studie berichten Wissenschaftler:innen aus dem Labor von Emmanuel Saliba und anderen deutschen Forschungseinrichtungen, dass Makrophagen – Fresszellen des Immunsystems, die Fremdstoffe unschädlich machen – dabei eine zentrale Rolle spielen. Einige Mechanismen, die an der infektionsbedingten Ateminsuffizienz beteiligt sind, ähneln denen der idiopathischen Lungenfibrose, einer bisher unheilbaren Krankheit, die zur Vernarbung der Lunge führt. Fehlgeleitete Wundheilungsreaktionen, die zur Vernarbung führen, könnten erklären, warum die Lunge lange funktionsunfähig bleibt und eine langwierige ECMO-Therapie erfordert.
Publikationen
2024
Human cytomegalovirus exploits STING signaling and counteracts IFN/ISG induction to facilitate infection of dendritic cells
Costa B, Becker J, Krammer T, Mulenge F, Durán V, Pavlou A, Gern OL, Chu X, Li Y, Cicin-Šain L, …, Erhard F, Kalinke U (2024)
Nature Communications 15 (1): 1745
Circulating NK cells establish tissue residency upon acute infection of skin and mediate accelerated effector responses to secondary infection
Torcellan T, Friedrich C, Doucet-Ladevèze R, Ossner T, Solé VV, Riedmann S, Ugur M, Imdahl F, Rosshart SP, Arnold SJ, …, Kastenmüller W, Gasteiger G (2024)
Immunity 57 (1): 124-140.e7
2023
Sequential Antigen-loss and Branching Evolution in Lymphoma after CD19- and CD20-Targeted T-cell Redirecting Therapy
Duell J, Leipold AM, Appenzeller S, Fuhr V, Rauert-Wunderlich H, Da Vià MC, Dietrich O, Toussaint C, Imdahl F, Eisele F, …, Saliba AE, Rasche L (2023)
Blood 143 (8): 685-696
Brain-to-gut trafficking of alpha-synuclein by CD11c+ cells in a mouse model of Parkinson's disease
McFleder RL, Makhotkina A, Groh J, Keber U, Imdahl F, Peña Mosca J, Peteranderl A, Wu J, Tabuchi S, Hoffmann J, …, Volkmann J, Ip CW (2023)
Nature Communications 14 (1): 7529
Vector-borne Trypanosoma brucei parasites develop in artificial human skin and persist as skin tissue forms
Reuter C, Hauf L, Imdahl F, Sen R, Vafadarnejad E, Fey P, Finger T, Jones NG, Walles H, Barquist L, …, Groeber-Becker F, Engstler M (2023)
Nature Communications 14 (1): 7660
Microglia-mediated demyelination protects against CD8+ T cell-driven axon degeneration in mice carrying PLP defects
Groh J, Abdelwahab T, Kattimani Y, Hörner M, Loserth S, Gudi V, Adalbert R, Imdahl F, Saliba AE, Coleman M, …, Simons M, Martini R (2023)
Nature Communications 14 (1): 6911
Th17.1 cell driven sarcoidosis-like inflammation after anti-BCMA CAR T cells in multiple myeloma
Leipold AM, Werner RA, Düll J, Jung P, John M, Stanojkovska E, Zhou X, Hornburger H, Ruckdeschel A, Dietrich O, …, Saliba AE, Rasche L (2023)
Leukemia 37 (3): 650-658
Myocardial Milieu Favors Local Differentiation of Regulatory T Cells
Delgobo M, Weiß E, ElDin Ashour D, Richter L, Popiolkowski L, Arampatzi P, Stangl V, Arias-Loza P, Mariotti-Ferrandiz E, Rainer PP, …, Frantz S, Campos Ramos G (2023)
Circulation Research 132 (5): 565-582
Integrated single-cell analysis based classification of vascular mononuclear phagocytes in mouse and human atherosclerosis
Zernecke A, Erhard F, Weinberger T, Schulz C, Ley K, Saliba AE, Cochain C (2023)
Cardiovascular Research 119 (8): 1676-1689
Dynamics of monocyte-derived macrophage diversity in experimental myocardial infarction
Rizzo G, Gropper J, Piollet M, Vafadarnejad E, Rizakou A, Bandi SR, Arampatzi P, Krammer T, DiFabion N, Dietrich O, …, Saliba AE, Cochain C (2023)
Cardiovascular Research 119 (3): 772-785
Rapid neutrophil mobilisation by VCAM-1+ endothelial extracellular vesicles
Akbar N, Braithwaite AT, Corr EM, Koelwyn GJ, van Solingen C, Cochain C, Saliba AE, Corbin A, Pezzolla D, Møller Jørgensen M, …, Melling GE, Shanmuganathan M (2023)
Cardiovascular Research 119 (1): 236-251
Genetic inhibition of CARD9 accelerates the development of atherosclerosis in mice through CD36 dependent-defective autophagy
Zhang Y, Vandestienne M, Lavillegrand JR, Joffre J, Santos-Zas I, Lavelle A, Zhong X, Le Goff W, Guérin M, Al-Rifai R, …, Sokol H, Ait-Oufella H (2023)
Nature Communications 14 (1): 4622
Short-range interactions between fibrocytes and CD8+ T cells in COPD bronchial inflammatory response
Eyraud E, Maurat E, Sac-Epée JM, Henrot P, Zysman M, Esteves P, Trian T, Dupuy JW, Leipold A, Saliba AE, …, Berger P, Dupin I (2023)
eLife 12: RP85875
Lymph node medulla regulates the spatiotemporal unfolding of resident dendritic cell networks
Ugur M, Labios RJ, Fenton C, Knöpper K, Jobin K, Imdahl F, Golda G, Hoh K, Grafen A, Kaisho T, …, Bajénoff M, Kastenmüller W (2023)
Immunity 58 (8): 1778-1793.e10
Cytotoxic CNS-associated T cells drive axon degeneration by targeting perturbed oligodendrocytes in PLP1 mutant mice
Abdelwahab T, Stadler D, Knöpper K, Arampatzi P, Saliba AE, Kastenmüller W, Martini R, Groh J (2023)
iScience 26 (5): 106698
An interferon gamma response signature links myocardial aging and immunosenescence
Ashour D, Rebs S, Arampatzi P, Saliba AE, Dudek J, Schulz R, Hofmann U, Frantz S, Cochain C, Streckfuß-Bömeke K, Ramos GC (2023)
Cardiovascular Research 119 (14): 2458-2468
Advancing massive transcriptional profiling of single bacteria
Saliba AE (2023)
Cell Reports Methods 3 (2): 100416
A primary cell-based in vitro model of the human small intestine reveals host olfactomedin 4 induction in response to Salmonella Typhimurium infection
Däullary T, Imdahl F, Dietrich O, Hepp L, Krammer T, Fey C, Neuhaus W, Metzger M, Vogel J, Westermann AJ, Saliba AE, Zdzieblo D (2023)
Gut Microbes 15 (1): 2186109
Altered and allele-specific open chromatin landscape reveals epigenetic and genetic regulators of innate immunity in COVID-19
Zhang B, Zhang Z, Koeken VACM, Kumar S, Aillaud M, Tsay HC, Liu Z, Kraft ARM, Soon CF, Odak I, …, Schulte LN, Li Y (2023)
Cell Genomics 3 (2): 100232
A MATQ-seq-Based Protocol for Single-Cell RNA-seq in Bacteria
Homberger C, Saliba AE, Vogel J (2023)
Methods in Molecular Biology 2584: 105-121
2022
JAK2V617F mutation drives vascular resident macrophages toward a pathogenic phenotype and promotes dissecting aortic aneurysm
Al-Rifai R, Vandestienne M, Lavillegrand JR, Mirault T, Cornebise J, Poisson J, Laurans L, Esposito B, James C, Mansier O, …, Rautou PE, Ait-Oufella H (2022)
Nature Communications 13 (1): 6592
Postnatal expansion of mesenteric lymph node stromal cells towards reticular and CD34+ stromal cell subsets
Pezoldt J, Wiechers C, Zou M, Litovchenko M, Biocanin M, Beckstette M, Sitnik K, Palatella M, van Mierlo G, Chen W, …, Deplancke B, Huehn J (2022)
Nature Communications 13 (1): 7227
Helicobacter pylori shows tropism to gastric differentiated pit cells dependent on urea chemotaxis
Aguilar C, Pauzuolis M, Pompaiah M, Vafadarnejad E, Arampatzi P, Fischer M, Narres D, Neyazi M, Kayisoglu Ö, Sell T, …, Saliba AE, Bartfeld S (2022)
Nature Communications 13 (1): 5878
Fibroblastic reticular cells mitigate acute graft-versus-host disease via MHCII-dependent maintenance of regulatory T cells
Shaikh H, Pezoldt J, Mokhtari Z, Gamboa Vargas J, Le DD, Peña Mosca J, Arellano-Viera E, Kern MA, Graf C, Beyersdorf N, …, Huehn J, Beilhack A (2022)
JCI Insight 7 (22): e154250
Landscape and age dynamics of immune cells in the Egyptian rousette bat
Friedrichs V, Toussaint C, Schäfer A, Rissmann M, Dietrich O, Mettenleiter TC, Pei G, Balkema-Buschmann A, Saliba AE, Dorhoi A (2022)
Cell Reports 40 (10): 111305
Nonproductive exposure of PBMCs to SARS-CoV-2 induces cell-intrinsic innate immune responses
Kazmierski J, Friedmann K, Postmus D, Emanuel J, Fischer C, Jansen J, Richter A, Bosquillon de Jarcy L, Schüler C, Sohn M, …, Niemeyer D, Goffinet C (2022)
Molecular Systems Biology 18 (8): e10961
Lymphatic migration of unconventional T cells promotes site-specific immunity in distinct lymph nodes
Ataide MA, Knöpper K, Cruz de Casas P, Ugur M, Eickhoff S, Zou M, Shaikh H, Trivedi A, Grafen A, Yang T, …, Gasteiger G, Kastenmüller W (2022)
Immunity 55 (10): 1813–1828.e9
Complement activation induces excessive T cell cytotoxicity in severe COVID-19
Georg P, Astaburuaga-García R, Bonaguro L, Brumhard S, Michalick L, Lippert LJ, Kostevc T, Gäbel C, Schneider M, Streitz M, …, Sawitzki B, PA-COVID-19 Study Group (2022)
Cell 185 (3): 493-512.e25
Interleukin-23 receptor expressing γδ T cells locally promote early atherosclerotic lesion formation and plaque necrosis in mice
Gil-Pulido J, Amézaga N, Jorgacevic I, Manthey HD, Rösch M, Brand T, Cidlinsky P, Schäfer S, Beilhack A, Saliba AE, …, Cochain C, Zernecke A (2022)
Cardiovascular Research 118 (14): 2932-2945
Time-resolved single-cell RNA-seq using metabolic RNA labelling
Erhard F, Saliba AE, Lusser A (2022)
Nature Reviews Methods Primers 2 (1): 331
Type 1 conventional dendritic cells maintain and guide the differentiation of precursors of exhausted T cells in distinct cellular niches
Dähling S, Mansilla AM, Knöpper K, Grafen A, Utzschneider DT, Ugur M, Whitney PG, Bachem A, Arampatzi P, Imdahl F, …, Bedoui S, Kastenmüller W (2022)
Immunity 55 (4): 656-670.e8
2021
Effector differentiation downstream of lineage commitment in ILC1s is driven by Hobit across tissues
Friedrich C, Taggenbrock RLRE, Doucet-Ladevèze R, Golda G, Moenius R, Arampatzi P, Kragten NAM, Kreymborg K, Gomez de Agüero M, Kastenmüller W, …, van Gisbergen KPJM, Gasteiger G (2021)
Nature Immunology 22 (10): 1256-1267
SARS-CoV-2 infection triggers profibrotic macrophage responses and lung fibrosis
Wendisch D, Dietrich O, Mari T, von Stillfried S, Ibarra IL, Mittermaier M, Mache C, Chua RL, Knoll R, Timm S, …, Saliba AE, Sander LE (2021)
Cell 184 (26): 6243-6261.e27
Homozygous BCMA gene deletion in response to anti-BCMA CAR T cells in a patient with multiple myeloma
Da Vià MC, Dietrich O, Truger M, Arampatzi P, Duell J, Heidemeier A, Zhou X, Danhof S, Kraus S, Chatterjee M, …, Saliba AE, Rasche L (2021)
Nature Medicine 27 (4): 616-619
Opposing Wnt signals regulate cervical squamocolumnar homeostasis and emergence of metaplasia
Chumduri C, Gurumurthy RK, Berger H, Dietrich O, Kumar N, Koster S, Brinkmann V, Hoffmann K, Drabkina M, Arampatzi P, …, Saliba AE, Meyer TF (2021)
Nature Cell Biology 23 (2): 184–197
Dysregulated Immunometabolism Is Associated with the Generation of Myeloid-Derived Suppressor Cells in Staphylococcus aureus Chronic Infection
Dietrich O, Heinz A, Goldmann O, Geffers R, Beineke A, Hiller K, Saliba AE, Medina E (2021)
Journal of Innate Immunity: 1-18
The healing myocardium mobilises a distinct B-cell subset through a CXCL13-CXCR5-dependent mechanism
Heinrichs M, Ashour D, Siegel J, Büchner L, Wedekind G, Heinze KG, Arampatzi P, Saliba AE, Cochain C, Hofmann U, Frantz S, Campos Ramos G (2021)
Cardiovascular Research 117 (13): 2664-2676
Time-Resolved scRNA-Seq Tracks the Adaptation of a Sensitive MCL Cell Line to Ibrutinib Treatment
Fuhr V, Vafadarnejad E, Dietrich O, Arampatzi P, Riedel A, Saliba AE, Rosenwald A, Rauert-Wunderlich H (2021)
International Journal of Molecular Sciences 22 (5): 2276
Initial HCV infection of adult hepatocytes triggers a temporally structured transcriptional program containing diverse pro- and anti-viral elements
Tegtmeyer B, Vieyres G, Todt D, Lauber C, Ginkel C, Engelmann M, Herrmann M, Pfaller CK, Vondran FW, Broering R, …, Pietschmann T, Brown RJ (2021)
Journal of Virology 95 (10)
A genome-wide transcriptomic analysis of embryos fathered by obese males in a murine model of diet-induced obesity
Bernhardt L, Dittrich M, El-Merahbi R, Saliba AE, Müller T, Sumara G, Vogel J, Nichols-Burns S, Mitchell M, Haaf T, El Hajj N (2021)
Scientific Reports 11: 1979
2020
Longitudinal Multi-omics Analyses Identify Responses of Megakaryocytes, Erythroid Cells, and Plasmablasts as Hallmarks of Severe COVID-19
Bernardes JP, Mishra N, Tran F, Bahmer T, Best L, Blase JI, Bordoni D, Franzenburg J, Geisen U, Josephs-Spaulding J, …, Schultze JL, Rosenstiel P (2020)
Immunity 53 (6): 1296-1314.e9
Dynamics of Cardiac Neutrophil Diversity in Murine Myocardial Infarction
Vafadarnejad E, Rizzo G, Krampert L, Arampatzi P, Arias-Loza A, Nazzal Y, Rizakou A, Knochenhauer T, Bandi SR, Nugroho VA, …, Saliba AE, Cochain C (2020)
Circulation Research 127 (9): e232-249
LifeTime and improving European healthcare through cell-based interceptive medicine
Rajewsky N, Almouzni G, Gorski SA, Aerts S, Amit I, Bertero MG, Bock C, Bredenoord AL, Cavalli G, Chiocca S, …, Vidal M, Voet T (2020)
Nature 587 (7834): 377-386
Single-cell RNA-sequencing reports growth-condition-specific global transcriptomes of individual bacteria
Imdahl F, Vafadarnejad E, Homberger C, Saliba AE, Vogel J (2020)
Nature Microbiology 5 (10): 1202–1206
Severe COVID-19 is marked by a dysregulated myeloid cell compartment
Schulte-Schrepping J, Reusch N, Paclik D, Baßler K, Schlickeiser S, Zhang B, Krämer B, Krammer T, Brumhard S, Bonaguro L, …, Saliba AE, Sander LE (2020)
Cell 182 (6): 1419-1440
Eleven grand challenges in single-cell data science
Lähnemann D, Köster J, Szczurek E, McCarthy DJ, Hicks SC, Robinson MD, Vallejos CA, Campbell KR, Beerenwinkel N, Mahfouz A, …, Shah SP, Schönhuth A (2020)
Genome Biology 21: 31
Advances and challenges in single-cell RNA-seq of microbial communities
Imdahl F, Saliba A (2020)
Current Opinion in Microbiology 57: 102-110
Tracheal brush cells release acetylcholine in response to bitter tastants for paracrine and autocrine signaling
Hollenhorst MI, Jurastow I, Nandigama R, Appenzeller S, Li L, Vogel J, Wiederhold S, Althaus M, Empting M, Altmüller J, …, Saliba AE, Krasteva-Christ G (2020)
The FASEB Journal 34 (1): 316-332
2019
scSLAM-seq reveals core features of transcription dynamics in single cells
Erhard F, Baptista MA, Krammer T, Hennig T, Lange M, Arampatzi P, Jürges CS, Theis FJ, Saliba AE, Dölken L (2019)
Nature 571 (7765): 419-423
2018
Salmonella persisters undermine host immune defenses during antibiotic treatment
Stapels DA, Hill PW, Westermann AJ, Fisher RA, Thurston TL, Saliba AE, Blommestein I, Vogel J, Helaine S (2018)
Science 362 (6419): 1156-1160
Neonatally imprinted stromal cell subsets induce tolerogenic dendritic cells in mesenteric lymph nodes
Pezoldt J, Pasztoi M, Zou M, Wiechers C, Beckstette M, Thierry GR, Vafadarnejad E, Floess S, Arampatzi P, Buettner M, …, Saliba AE, Huehn J (2018)
Nature Communications 9: 3903
Single-Cell RNA-Seq Reveals the Transcriptional Landscape and Heterogeneity of Aortic Macrophages in Murine Atherosclerosis
Cochain C, Vafadarnejad E, Arampatzi P, Pelisek J, Winkels H, Ley K, Wolf D, Saliba AE, Zernecke A (2018)
Circulation Research 122 (12): 1661-1674
Atlas of the Immune Cell Repertoire in Mouse Atherosclerosis Defined by Single-Cell RNA-Sequencing and Mass Cytometry
Winkels H, Ehinger E, Vassallo M, Buscher K, Dinh HQ, Kobiyama K, Hamers AA, Cochain C, Vafadarnejad E, Saliba AE, …, Ley K, Wolf D (2018)
Circulation Research 122 (12): 1675-1688
Genome organization and DNA accessibility control antigenic variation in trypanosomes
Müller LS, Cosentino RO, Förstner KU, Guizetti J, Wedel C, Kaplan N, Janzen CJ, Arampatzi P, Vogel J, Steinbiss S, …, Sebra RP, Siegel TN (2018)
Nature 563 (7729): 121-125
Tolerogenic Transcriptional Signatures of Steady-State and Pathogen-Induced Dendritic Cells
Vendelova E, Ashour D, Blank P, Erhard F, Saliba AE, Kalinke U, Lutz MB (2018)
Frontiers in Immunology 9: 333
2017
Einzelzell-RNA-Sequenzierung beleuchtet den Infektionsprozess
Saliba AE, Westermann AJ, Vogel J (2017)
BIOspektrum 23 (5): 525-528
New RNA-seq approaches for the study of bacterial pathogens
Saliba AE, Santos SC, Vogel J (2017)
Current Opinion in Microbiology 35: 78-87
2016
A protocol for the systematic and quantitative measurement of protein-lipid interactions using the liposome-microarray-based assay
Saliba AE, Vonkova I, Deghou S, Ceschia S, Tischer C, Kugler KG, Bork P, Ellenberg J, Gavin A (2016)
Nature Protocols 11 (6): 1021-38
Single-cell RNA-seq ties macrophage polarization to growth rate of intracellular Salmonella
Saliba AE, Li L, Westermann AJ, Appenzeller S, Stapels DA, Schulte LN, Helaine S, Vogel J (2016)
Nature Microbiology 2: 16206
2015
The systematic analysis of protein-lipid interactions comes of age
Saliba AE, Vonkova I, Gavin A (2015)
Nature Reviews Molecular Cell Biology 16 (12): 753-61
Lipid Cooperativity as a General Membrane-Recruitment Principle for PH Domains
Vonkova I, Saliba AE, Deghou S, Anand K, Ceschia S, Doerks T, Galih A, Kugler KG, Maeda K, Rybin V, …, Bork P, Gavin A (2015)
Cell Reports 12 (9): 1519-30
2014
A quantitative liposome microarray to systematically characterize protein-lipid interactions
Saliba AE, Vonkova I, Ceschia S, Findlay GM, Maeda K, Tischer C, Deghou S, van Noort V, Bork P, Pawson T, Ellenberg J, Gavin A (2014)
Nature Methods 11 (1): 47-50
Single-cell RNA-seq: advances and future challenges
Saliba AE, Westermann AJ, Gorski SA, Vogel J (2014)
Nucleic Acids Research 42 (14): 8845-60
2010
Microfluidic sorting and multimodal typing of cancer cells in self-assembled magnetic arrays
Saliba AE, Saias L, Psychari E, Minc N, Simon D, Bidard F, Mathiot C, Pierga J, Fraisier V, Salamero J, …, Malaquin L, Viovy J (2010)
PNAS 107 (33): 14524-9
2009
Cellules tumorales circulantes et cancer du sein : méthodes de détection et résultats cliniques
Bidard F, Saliba AE, Saias L, Degeorges A, Cremoux Pd, Viovy J, Vincent-Salomon A, Mathiot C, Pierga J, Gramont A (2009)
Bulletin du Cancer 96 (1): 73-86
2004
Nanotechnology serving biochips – The Toulouse example
Vieu C, Malaquin L, Thibault C, Saliba AE, Daran E, Dildan M, Carcenac F, Leberre V, Trevisiol E, François JM (2004)
Biofutur (250): 41-45