Jun.-Prof. Jens Hör
Molekulare Grundlagen von RNA-Phagen
Unsere Forschung
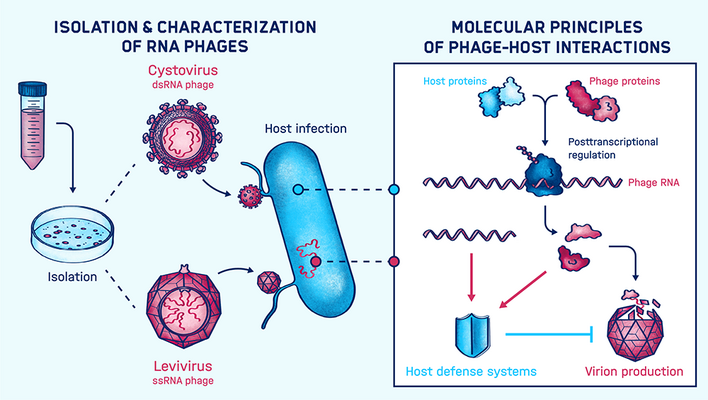
Die Arbeitsgruppe von Jens Hör untersucht die Mechanismen der Infektion, Wirtsübernahme und Phagenabwehr im Rahmen der Interaktion von RNA-Phagen mit ihren Wirten. Ihr Ziel ist es, die molekularen Prinzipien zu ergründen, die diesen Prozessen zugrunde liegen, um neue und verbesserte antibakterielle Behandlungsstrategien, wie beispielsweise die Phagentherapie, zu entwickeln.
Angesichts der zunehmenden Bedrohung durch antibiotikaresistente Bakterien sind neue Behandlungsmöglichkeiten für bakterielle Infektionen dringend erforderlich. Phagen als die natürlichen Gegenspieler von Bakterien könnten Abhilfe schaffen. Hierfür ist es jedoch entscheidend, geeignete Phagen zu identifizieren und ihre Funktionsweise zu entschlüsseln. RNA-Phagen sind – im Gegensatz zu DNA-Phagen – wenig erforscht. Ihre Charakterisierung wird dazu beitragen, das volle Potenzial von Phagen bei der Entwicklung neuartiger antimikrobieller Therapien auszuschöpfen und birgt die Chance, innovative molekulare Werkzeuge und Technologien zu konzipieren.
Die Arbeitsgruppe von Jens Hör untersucht die molekularen Grundlagen dieser RNA-Phagen im Kontext der Phagen-Wirt-Interaktionen. Ihre Forschung zielt darauf ab, zu ergründen, wie RNA-Phagen ihre Wirte während der Infektion unter ihre Kontrolle bringen und wie die Wirte sich gegen die eindringenden Phagen verteidigen. Außerdem isoliert das Hör-Labor neue RNA-Phagen und charakterisiert sie funktionell.
Die Gruppe nutzt sowohl klassische biochemische und molekularbiologische Methoden als auch modernste Hochdurchsatzverfahren, um die besondere Lebensweise von RNA-Phagen im Zusammenspiel mit ihren Wirten zu analysieren. Übergeordnetes Ziel ist es, RNA-Phagen besser zu verstehen und herauszufinden, wie sie für die Entwicklung neuer antibakterieller Strategien genutzt werden können.
Team-Mitglieder

Jun.-Prof. Jens Hör
Gruppenleiter

Manuel Velasco Gomariz
Postdoc

Aatreyi Roy
Doktorandin

Arina Drobysheva
Doktorandin

Sarah Reichardt
Technische Assistentin
Forschungsprojekte
Dem Virus ein Schnippchen schlagen
Phagen – Viren, die Bakterien befallen – bestehen aus einem Kopf und einem Schwanz. Der Kopf enthält das genetische Material des Phagen. Die Schwanzfasern dienen dazu, einen potenziellen Wirt zu finden, also eine Bakterienzelle, in die er dieses Material einbringen kann. Nach der Injektion übernimmt der Phage die Zellmaschinerie des Bakteriums: Er zwingt es, neue Phagen-Kopien zu produzieren. Diese Kopien sprengen schließlich die Zelle und infizieren andere Bakterien in der Kolonie. Jens Hör und Kolleg:innen vom Weizmann Institute of Science beschreiben in der Fachzeitschrift Nature ein bakterielles Immunsystem, das den Plan der Phagen durchkreuzt, indem es ein kleines Eiweißmolekül an ihren Schwanz bindet. Die Komponenten dieses neuen Immunsystems ähneln in ihrer Struktur einem menschlichen Immunmechanismus. Sie könnten dazu beitragen, dessen Funktionsweise und somit die Entstehung unseres eigenen Immunsystems besser zu beleuchten.
Publikationen
2025
2024
Bacteria conjugate ubiquitin-like proteins to interfere with phage assembly
Hör J, Wolf SG, Sorek R (2024)
Nature 631 (8022): 850-856
2023
Discovery of phage determinants that confer sensitivity to bacterial immune systems
Stokar-Avihail A, Fedorenko T, Hör J, Garb J, Leavitt A, Millman A, Shulman G, Wojtania N, Melamed S, Amitai G, Sorek R (2023)
Cell 186 (9): 1863-1876.e16
2022
INRI-seq enables global cell-free analysis of translation initiation and off-target effects of antisense inhibitors
Hör J, Jung J, Ðurica-Mitic S, Barquist L, Vogel J (2022)
Nucleic Acids Research 50 (22): e128
An overview of gene regulation in bacteria by small RNAs derived from mRNA 3' ends
Ponath F, Hör J, Vogel J (2022)
FEMS Microbiology Reviews 46 (5): fuac017
An expanded arsenal of immune systems that protect bacteria from phages
Millman A, Melamed S, Leavitt A, Doron S, Bernheim A, Hör J, Garb J, Bechon N, Brandis A, Lopatina A, …, Amitai G, Sorek R (2022)
Cell Host & Microbe 30 (11): 1556-1569.e5
Global profiling of the RNA and protein complexes of Escherichia coli by size exclusion chromatography followed by RNA sequencing and mass spectrometry (SEC-seq)
Chihara K, Gerovac M, Hör J, Vogel J (2022)
RNA 29 (1): 123-39
2021
Analysis of the RNA and Protein Complexome by Grad-seq
Hör J, Vogel J (2021)
Methods in Molecular Biology 2300: 183-201
Grad-seq identifies KhpB as a global RNA-binding protein in Clostridioides difficile that regulates toxin production
Lamm-Schmidt V, Fuchs M, Sulzer J, Gerovac M, Hör J, Dersch P, Vogel J, Faber F (2021)
microLife 2: uqab004
2020
Grad-seq shines light on unrecognized RNA and protein complexes in the model bacterium Escherichia coli
Hör J, Di Giorgio S, Gerovac M, Venturini E, Förstner KU, Vogel J (2020)
Nucleic Acids Research 48 (16): 9301-9319
Grad-seq in a Gram-positive bacterium reveals exonucleolytic sRNA activation in competence control
Hör J, Garriss G, Di Giorgio S, Hack LM, Vanselow JT, Förstner KU, Schlosser A, Henriques-Normark B, Vogel J (2020)
The EMBO Journal 39 (9): e103852
Trans-Acting Small RNAs and Their Effects on Gene Expression in Escherichia coli and Salmonella enterica
Hör J, Matera G, Vogel J, Gottesman S, Storz G (2020)
EcoSal Plus 9 (1): 10.1128/ecosalplus.ESP-0030-2019
2018
Bacterial RNA Biology on a Genome Scale
Hör J, Gorski SA, Vogel J (2018)
Molecular Cell 70 (5): 785-799
2017
A Small Number of Low-abundance Bacteria Dominate Plant Species-specific Responses during Rhizosphere Colonization
Dawson W, Hör J, Egert M, van Kleunen M, Pester M (2017)
Frontiers in Microbiology 8: 975
Discovery of new RNA classes and global RNA-binding proteins
Smirnov A, Schneider C, Hör J, Vogel J (2017)
Current Opinion in Microbiology 39: 152-160