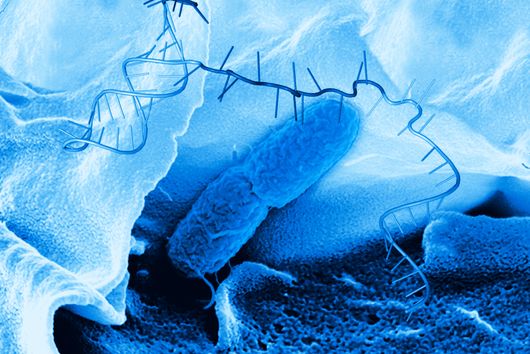
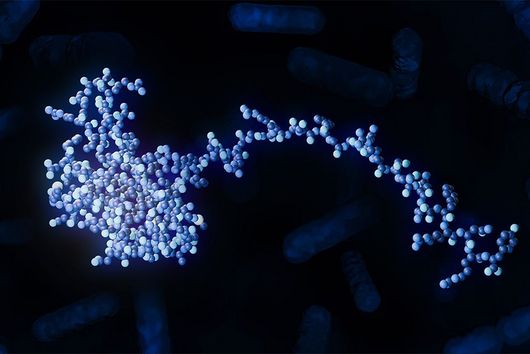
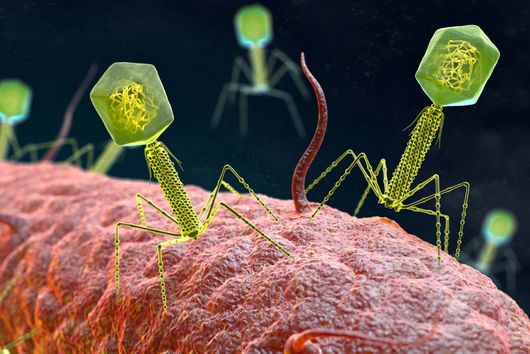
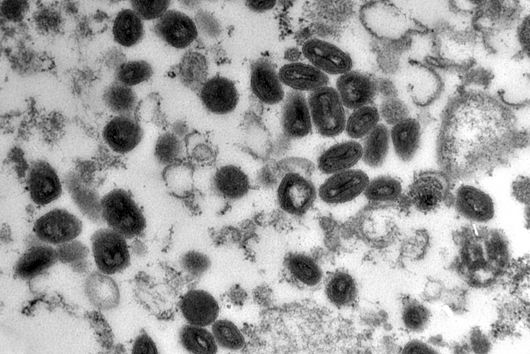
Phagen – Viren, die Bakterien befallen – bestehen aus einem Kopf und einem Schwanz. Der Kopf enthält das genetische Material des Phagen. Die Schwanzfasern dienen dazu, einen potenziellen Wirt zu finden, also eine Bakterienzelle, in die er dieses Material einbringen kann. Nach der Injektion übernimmt der Phage die Zellmaschinerie des Bakteriums: Er zwingt es, neue Phagen-Kopien zu produzieren. Diese Kopien sprengen schließlich die Zelle und infizieren andere Bakterien in der Kolonie. Eine neue Studie des Weizmann Institute of Science, die in der Fachzeitschrift Nature erschienen ist, beschreibt ein bakterielles Immunsystem, das den Plan der Phagen durchkreuzt, indem es ein kleines Eiweißmolekül an ihren Schwanz heftet. Die Komponenten dieses neuen Immunsystems ähneln in ihrer Struktur einem menschlichen Immunmechanismus. Sie könnten dazu beitragen, dessen Funktionsweise und somit die Entstehung unseres eigenen Immunsystems besser zu beleuchten. Jens Hör, der das Forschungsprojekt als Postdoc im Labor von Rotem Sorek am Weizmann-Institut durchgeführt hat, ist mittlerweile Gruppenleiter am Helmholtz-Institut Würzburg (HIRI).
Die ersten Antiphagen-Abwehrmechanismen in Bakterien wurden zwar bereits in den 1960er-Jahren entdeckt, doch bis vor kurzem waren nur wenige von ihnen bekannt. Das wohl prominenteste Abwehrsystem ist CRISPR-Cas9, dessen Entdeckung zu einer Revolution in der Genom-Editierung geführt hat. In den vergangenen Jahren gab es jedoch eine Flut neuer Erkenntnisse auf diesem Gebiet, die zur Beschreibung von mehr als 150 bakteriellen Immunsystemen mit unterschiedlichen Wirkmechanismen beigetragen haben. Viele dieser Systeme wurden mithilfe einer Methode identifiziert, die Rotem Sorek von der Abteilung für Molekulargenetik am Weizmann-Institut entwickelt hat.
Soreks Methode beruht dabei auf einem bestechend einfachen Prinzip: Gene, die an bakteriellen Immunmechanismen beteiligt sind, neigen dazu, sich im Bakteriengenom in bestimmten Bereichen zu sammeln – sie bilden also sogenannte „Abwehrinseln“. Forschende können daher neue Immunsysteme entdecken, indem sie Gene mit unbekannter Funktion untersuchen, die sich in der Nähe solcher bekannten Abwehrinseln befinden. „In vielen unserer Studien haben wir Komponenten des bakteriellen Immunsystems ausgemacht, die wir bereits von umfassend untersuchten menschlichen Immunmechanismen her kannten“, erklärt Sorek. „Das deutet darauf hin, dass ein großer Teil unseres angeborenen Immunsystems seinen evolutionären Ursprung in Bakterien hat. Unsere neue Studie unterstützt diese Hypothese.”
Ubiquitin auf den Schultern von Giganten
In den 1970er-Jahren entdeckten Wissenschaftler:innen ein zelluläres Kontrollsystem, das die Struktur, Funktion und Lebensdauer von Proteinen verändern kann, indem es ein kleines Protein mit der Bezeichnung Ubiquitin an sie bindet. Seit der Entdeckung von Ubiquitin – für die die Professoren Aaron Ciechanover, Avram Hershko und Irwin Rose 2004 den Nobelpreis für Chemie erhielten – haben Forschende viele ähnliche Systeme entdeckt, bei denen Enzyme verschiedene kleine Proteine an das Zielprotein binden und so dessen Verbleib verändern.
In der Studie entdeckte ein Forschungsteam um Jens Hör ein neues bakterielles Immunsystem, das ein Ubiquitin-ähnliches Protein enthält. Das Protein gleicht in seiner Struktur ISG15, einem der rätselhaftesten Proteine des menschlichen Immunsystems. ISG15 spielt eine Rolle bei der Abwehr verschiedener Viren wie Influenza und HIV – wie es diese Aufgabe erfüllt, ist jedoch noch nicht vollständig verstanden.
Hör und Kolleg:innen stellten fest, dass das von ihnen entdeckte System im Gegensatz zu anderen bakteriellen Immunsystemen Viren nicht daran hindert, die Zelle zu kapern und Duplikate von sich selbst zu produzieren: Bakterien, die dieses Immunsystem kodierten, starben nach einer Infektion und erzeugten neue Virusnachkommen. Diese Viren waren jedoch „steril“, das heißt, sie konnten keine anderen Bakterien infizieren. Daraus schlossen die Forschenden, dass das neue Immunsystem in der Lage sein muss, die Ausbreitung des Virus auf andere Zellen in der Kolonie zu verhindern.
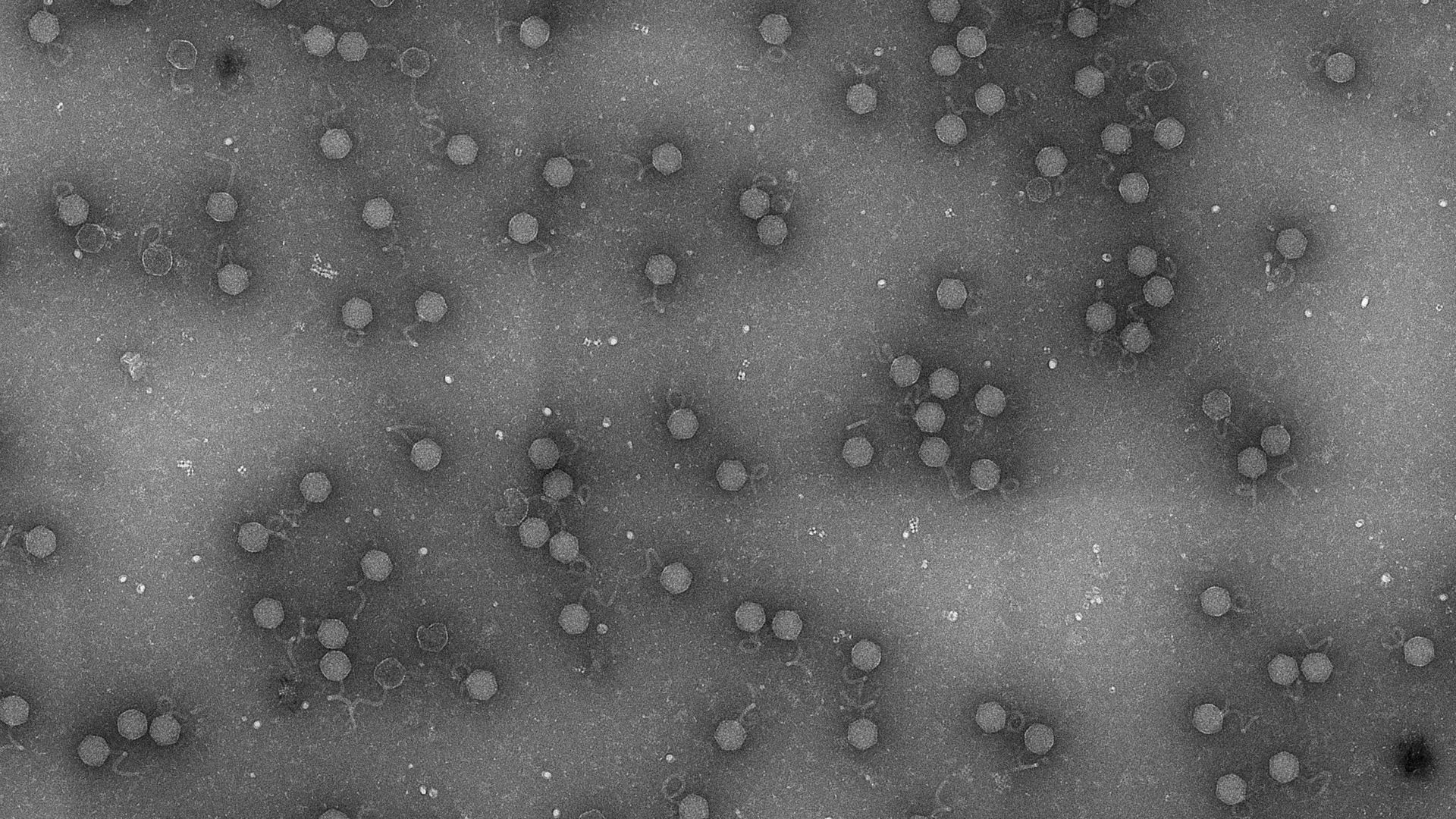
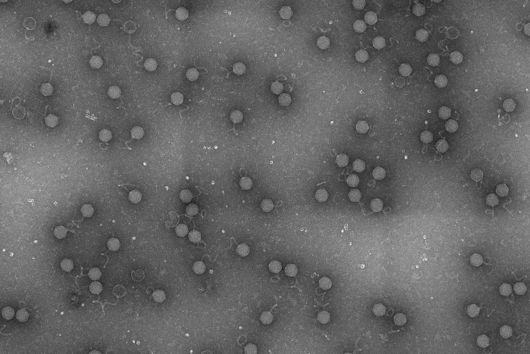
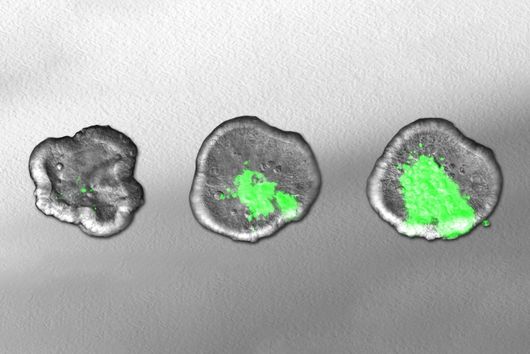
Um zu verstehen, warum die Virusnachkommen ihre Fähigkeit verlieren, andere Zellen zu infizieren, und welche Rolle das neue bakterielle Immunsystem dabei spielt, hat sich Soreks Forschungsteam mit Sharon Wolf zusammengetan, der Leiterin des Bereichs Elektronenmikroskopie am Weizmann-Institut. Die Forschenden markierten das Ubiquitin-ähnliche Protein im Zentrum des neuen Immunsystems mit Goldpartikeln, die unter dem Mikroskop deutlich sichtbar sind.
Als sie sich die Bilder der vermehrten Phagen ansahen, waren sie überrascht: Das markierte Protein saß am Ende des Virenschwanzes und hinderte die Phagen daran, mit ihren Schwanzfasern neue Bakterienzellen zu finden und zu infizieren. Die Forschenden gehen davon aus, dass das neue Immunsystem in der Lage ist, die dreidimensionale Struktur des Virenschwanzes zu erkennen, und so effektiv gegen eine Vielzahl von Phagen vorgehen kann – solange deren Schwanzfasern eine ähnliche Struktur aufweisen.
„Wir hoffen, dass unsere Entdeckung in Bakterien Forschende, die sich mit dem menschlichen Immunsystem beschäftigen, dazu inspiriert, zu untersuchen, ob ein ähnliches Prinzip auch für das menschliche Immunprotein ISG15 gilt. Viren, die den Menschen befallen, haben zwar keinen Schwanz, aber es ist durchaus möglich, dass die menschliche Abwehr auch durch die Störung eines wichtigen Strukturproteins des Virus funktioniert“, sagt Sorek. „Das Immunsystem, das wir in dieser Studie untersucht haben, ist nur eines von vielen, die Ubiquitin-ähnliche Proteine enthalten, die wir im bakteriellen Genom identifiziert haben. Es gilt nun abzuwarten, wie diese anderen Systeme ihre altbekannten Feinde, die Viren, bekämpfen.“
Text: Weizmann Institute of Science (angepasst durch das HIRI)

































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/5/4/csm_2024_10_EUREKA__c_HIRI_Luisa_Macharowsky_3_low_01_863006bd0e.jpg)































![[Translate to German:]](/fileadmin/HIRI/__processed__/3/3/csm_2022_07_BEISEL_Chase_2_cropped_Low_a7a341cf03.jpg)





































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/2/8/csm_Collage_WUE_9fa1cd516c.jpg)










![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/0/8/csm_05_Saliba_02_c_HIRI_Luisa_Macharowsky_ohne_Lacoste_low_zugeschnitten_2_01_9822d73a3f.jpg)






![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/c/csm_HIRI-Grundstseinlegung_Foto__c_Mario_Schmitt_79ad64af6d.jpg)













![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/2/csm_2023_04_Smyth_Paper_c_HIRI_Luisa_Macharowsky_2_low_84f993d581.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/e/2/csm_2022_11_HIRI_City_of_Wuerzburg_Child_Care_c_HIRI_Luisa_Macharowsky_2_low_6445b253eb.jpg)
![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/3/9/csm_HIRI_key_visual_CHurtig_606d992d74.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/f/0/csm_Ahsen_blue2_6e28a4ee6b.jpg)












![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/5/csm_2022_02_Marco_Olguin_HIRI-Luisa_Macharowsky_04_low_new_3dc837191c.jpg)


![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/3/c/csm_Joerg_Vogel_Portrait_-_photocredit_Mario_Schmitt__c__HIRI_5013b9643b.jpg)



![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/6/csm_RNA_Delivery_1902376336.jpg)
![[Translate to Deutsch:]](/fileadmin/HIRI/__processed__/7/c/csm_Caliskan_Kibe_Pekarek_105c7d1860.jpg)
![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/c/1/csm_N-Caliskan_M-Zimmer_Zoom_c93c493293.jpg)