Würzburg, 1. Februar 2024 – Das Zusammenspiel von Ribonukleinsäuren (RNAs, von engl. ribonucleic acids) und Proteinen ist nicht nur für die Aufrechterhaltung des zellulären Gleichgewichts von Bedeutung, sondern steht auch im Mittelpunkt des Kräftemessens zwischen Virus und Wirt. Bislang gab es jedoch keine Methode, um direkte Interaktionen einzelner RNA-Regionen global zu kartieren, ohne dafür die Ziel-RNA oder -zelle genetisch verändern zu müssen. Forschende des Helmholtz-Instituts Würzburg und des Broad Institute of MIT and Harvard in den USA haben nun ein wegweisendes Werkzeug entwickelt, das diese Hürde nimmt. Ihre Ergebnisse wurden kürzlich in der Fachzeitschrift Nucleic Acids Research veröffentlicht.
Wenn RNA-Viren, zu denen auch das Coronavirus SARS-CoV-2 gehört, unsere Zellen infizieren, nutzen sie regulatorische RNA-Elemente, um Proteine von Virus und Wirt für ihre Genexpression und Vervielfältigung zu rekrutieren. Diese Wechselwirkungen viraler RNAs und der in ihnen enthaltenen Steuerungselemente zu verstehen, ist daher der erste Schritt, um Schwachstellen im viralen Vermehrungsprozess aufzudecken und die Entwicklung neuer antiviraler Wirkstoffe zu erleichtern.
Die Methode
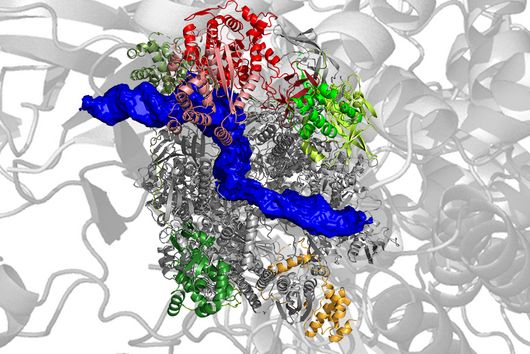
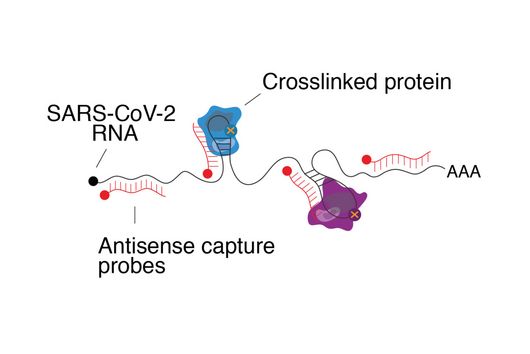
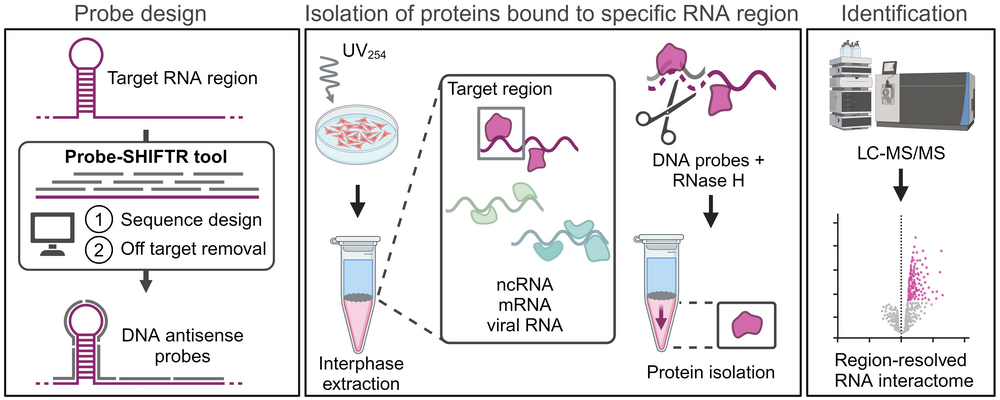
Forscher:innen des Helmholtz-Instituts für RNA-basierte Infektionsforschung (HIRI) in Würzburg haben jüngst in Zusammenarbeit mit dem Broad Institute of MIT and Harvard in den USA eine neue Methode entwickelt, die es erstmals ermöglicht, Interaktionen bestimmter Regionen innerhalb eines RNA-Moleküls zu ermitteln. Mit dem auf Massenspektrometrie basierenden Verfahren namens SHIFTR können Proteine, die mit einer speziellen RNA-Sequenz interagieren, umfassend kartiert werden – und das in lebenden Zellen und ohne gentechnische Veränderung.
„Bisher war es nicht möglich, das Zusammenspiel von Proteinen und einzelnen RNA-Abschnitten in intakten Zellen zu untersuchen, ohne die Ziel-RNA oder -zelle genetisch zu modifizieren. Unsere SHIFTR-Methode eröffnet uns endlich diese Möglichkeit und ist zudem einfach durchzuführen. SHIFTR erlaubt nicht nur die Betrachtung einzelner Regionen, sondern benötigt im Vergleich zu herkömmlichen Verfahren wesentlich weniger Ausgangsmaterial, ist in hohem Maße skalierbar und kostengünstig“, erklärt Mathias Munschauer, der eine Forschungsgruppe am Helmholtz-Institut Würzburg leitet, einem Standort des Braunschweiger Helmholtz-Zentrums für Infektionsforschung (HZI) in Kooperation mit der Julius-Maximilians-Universität (JMU) Würzburg.
„Mit diesem neuen Werkzeug können wir die Interaktionen für praktisch jede zelluläre RNA und jedes regulatorische Element innerhalb dieser RNAs bestimmen“, freut sich Jens Aydin, Doktorand in der Arbeitsgruppe von Mathias Munschauer und Erstautor der Studie in Nucleic Acids Research. „Das kann die Art und Weise, wie wir die RNA einer Zelle betrachten, grundlegend verändern – ein wichtiger Meilenstein“, betont Munschauer.
SARS-CoV-2 im Visier
Mithilfe der neuen Methode konnte das Forschungsteam den Vermehrungsprozess von SARS-CoV-2 weiter beleuchten: Die Wissenschaftler:innen untersuchten verschiedene Sequenzelemente in SARS-CoV-2-RNAs, die während einer Infektion produziert werden. Zum ersten Mal gelang es ihnen, die Interaktionen an den 5'- und 3'-Endregionen der viralen RNA zu erfassen. Diese Bereiche enthalten bekanntermaßen regulatorische Elemente, die die Proteinproduktion, die RNA-Stabilität und die Replikation des viralen Genoms kontrollieren. Zusätzlich zu den bereits bekannten Akteuren entdeckten sie bisher noch nicht beobachtete Interaktionen mit Proteinen, die an der Synthese viraler RNAs beteiligt sind. Einige dieser neu entdeckten Wechselwirkungen könnten als Angriffsziele für neuartige antivirale Therapien in Frage kommen.
In Zukunft könnte SHIFTR eingesetzt werden, um die Regulation zellulärer Transkriptome, also der Gesamtheit aller RNA-Moleküle einer Zelle, in Gesundheit und Krankheit besser zu verstehen. Dies könnte neue Einblicke in regulatorische Abhängigkeiten und potenzielle Angriffspunkte für Medikamente liefern. Die Plattform ermöglicht es, die Wechselwirkung von RNA-basierten Therapeutika mit der Genexpressionsmaschinerie der Zielzelle genauer zu analysieren. Diese Erkenntnisse könnten als Grundlage für die Entwicklung optimierter RNA-basierter Medikamente, wie beispielsweise mRNA-Impfstoffe, dienen.
Förderung
Die Studie wurde aus Mitteln des Helmholtz Young Investigator Group-Programms, des Europäischen Forschungsrats (ERC) und des Forschungsnetzwerks FOR-COVID unterstützt. Nora Schmidt wurde außerdem über das EMBO Long-Term Fellowship-Programm gefördert. Die Helmholtz-Gemeinschaft hat die Open-Access-Gebühr finanziert.
Originalpublikation
Aydin J, Gabel A, Zielinski S, Ganskih S, Schmidt N, Hartigan CR, Schenone M, Carr SA, Munschauer M
SHIFTR enables the unbiased identification of proteins bound to specific RNA regions in live cells
Nucleic Acids Research (2024), DOI: https://doi.org/10.1093/nar/gkae038



![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/2/a/csm_2024_05_Infection___Immunity_Poster_Webdatei_c3325f947c.png)











![[Translate to German:]](/fileadmin/HIRI/__processed__/3/3/csm_2022_07_BEISEL_Chase_2_cropped_Low_a7a341cf03.jpg)




































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/2/8/csm_Collage_WUE_9fa1cd516c.jpg)










![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/0/8/csm_05_Saliba_02_c_HIRI_Luisa_Macharowsky_ohne_Lacoste_low_zugeschnitten_2_01_9822d73a3f.jpg)





![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/c/csm_HIRI-Grundstseinlegung_Foto__c_Mario_Schmitt_79ad64af6d.jpg)













![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/2/csm_2023_04_Smyth_Paper_c_HIRI_Luisa_Macharowsky_2_low_84f993d581.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/e/2/csm_2022_11_HIRI_City_of_Wuerzburg_Child_Care_c_HIRI_Luisa_Macharowsky_2_low_6445b253eb.jpg)
![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/3/9/csm_HIRI_key_visual_CHurtig_606d992d74.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/f/0/csm_Ahsen_blue2_6e28a4ee6b.jpg)












![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/5/csm_2022_02_Marco_Olguin_HIRI-Luisa_Macharowsky_04_low_new_3dc837191c.jpg)


![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/3/c/csm_Joerg_Vogel_Portrait_-_photocredit_Mario_Schmitt__c__HIRI_5013b9643b.jpg)



![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/6/csm_RNA_Delivery_1902376336.jpg)
![[Translate to Deutsch:]](/fileadmin/HIRI/__processed__/7/c/csm_Caliskan_Kibe_Pekarek_105c7d1860.jpg)
![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/c/1/csm_N-Caliskan_M-Zimmer_Zoom_c93c493293.jpg)