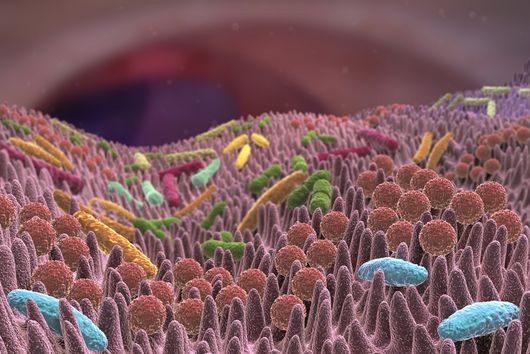
Würzburg, 11. Januar 2024 – Um Viren, Bakterien und andere Pathogene zu bekämpfen, bietet die synthetische Biologie neue technologische Ansätze, deren Effizienz in Experimenten validiert wird. Forschende vom Helmholtz-Institut Würzburg und der Kooperative Helmholtz AI haben mithilfe von Datenintegration und Künstlicher Intelligenz (KI) einen maschinellen Lernansatz entwickelt, der die Wirksamkeit von CRISPR-Technologien präziser als bisher vorhersagen kann. Die Erkenntnisse wurden heute im Fachmagazin Genome Biology publiziert.
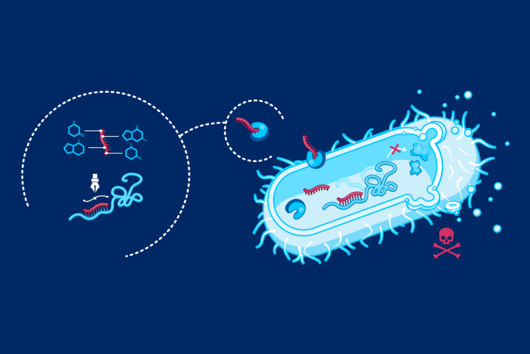
Im Erbgut – der DNA – eines Organismus steckt der Bauplan für Proteine und damit für die Herstellung neuer Zellen. Molekularbiologische CRISPR-Technologien ermöglichen es, einzelne Gene gezielt zu verändern oder abzuschalten und die Proteinproduktion zu unterbinden. Die Idee dahinter ist, Krankheitserreger zu bekämpfen, genetische Erkrankungen zu heilen oder andere nützliche Effekte zu erzielen.
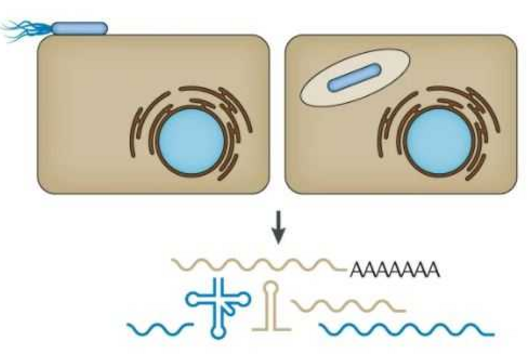
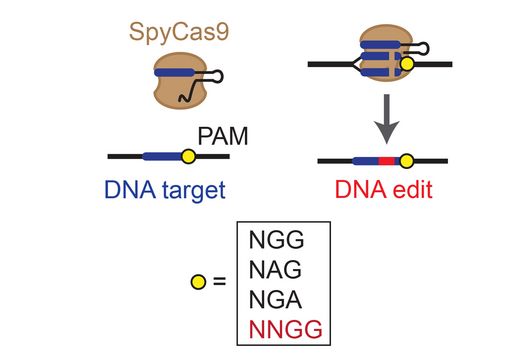
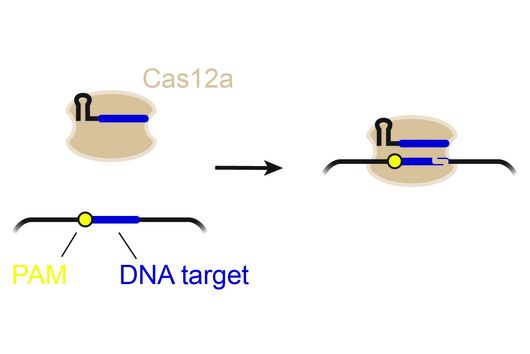
Eines dieser molekularbiologischen Werkzeuge ist CRISPRi (von “CRISPR-Interferenz”). Dabei handelt es sich um ein biochemisches Verfahren, das es ermöglicht, Gene und die Genexpression zu blockieren, ohne dass dafür die DNA-Sequenz verändert werden muss. Wie bei dem als „Genschere“ bekannten CRISPR-Cas-System steuert bei dieser Methode eine Ribonukleinsäure (RNA), die als Leit-RNA dient, eine Nuklease (Cas). Im Gegensatz zur Genschere bindet die Nuklease bei CRISPRi jedoch nur an spezifische Stellen der DNA, ohne diese zu schneiden. Die Bindung bewirkt, dass das entsprechende Gen nicht abgelesen wird und damit inaktiv bleibt.
Wie gut dieses Verfahren für ein bestimmtes Gen funktioniert, war bislang nur schwer vorherzusagen. Forschende vom Würzburger Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) in Zusammenarbeit mit der Universität Würzburg und der Kooperative Helmholtz AI (Helmholtz Artificial Intelligence Cooperation Unit) haben jetzt mithilfe von Datenintegration und Künstlicher Intelligenz (KI) einen maschinellen Lernansatz entwickelt, um solche Vorhersagen in Zukunft zu verbessern.
Der Ansatz
CRISPRi-Screens sind ein höchst empfindliches Werkzeug, mit dem Forschende die Effekte einer reduzierten Genexpression untersuchen können. In ihrer Studie, die heute im Fachmagazin Genome Biology erschienen ist, haben die Wissenschaftler:innen Daten aus mehreren genomweiten CRISPRi-Screens zusammengeführt, um einen maschinellen Lernansatz zu trainieren. Ihr Ziel: bessere Vorhersagen darüber, wie wirksam die im CRISPRi-System zum Einsatz kommenden, künstlich entworfenen Leit-RNAs sind.
„Genomweite Screens liefern leider nur indirekte Informationen über die Effizienz einer Leit-RNA. Deswegen haben wir eine neue maschinelle Lernmethode angewandt, die die Wirksamkeit der eingesetzten Leit-RNA getrennt vom Effekt des abgeschalteten Gens beleuchtet“, erläutert Lars Barquist. Der Computer-Biologe hat die Studie initiiert und leitet eine Bioinformatik-Forschungsgruppe am Würzburger Helmholtz-Institut, einem Standort des Braunschweiger Helmholtz-Zentrums für Infektionsforschung in Kooperation mit der Julius-Maximilians-Universität Würzburg.
Mithilfe weiterer KI-Werkzeuge stellte das Team verständliche Designregeln für künftige CRISPRi-Experimente auf. Ein umfassendes, auf essenzielle bakterielle Gene gerichtetes Screening, das die Studienautoren unabhängig von ihren ursprünglichen Trainingsdaten durchführten, validierte schließlich den Ansatz und ergab präzisere Prognosen.
„Die Ergebnisse haben gezeigt, dass unser Modell bisherige Methoden übertrifft und zuverlässigere Vorhersagen über die Leistung von CRISPRi bei der Ausrichtung auf bestimmte Gene liefert“, freut sich Yanying Yu, Doktorandin in der Arbeitsgruppe von Lars Barquist und Erstautorin der Studie.
Besonders überraschte die Forschenden, dass nicht die Leit-RNA selbst der wichtigste Faktor dafür ist zu bestimmen, wie gut diese in CRISPRi-Screens verringert wird. „Gewisse genspezifische Merkmale, die mit der Genexpression zusammenhängen, haben hierbei offenbar einen größeren Einfluss, als bisher angenommen“, erklärt Yu.
Zudem verdeutlicht die Studie, dass die Kombination von Informationen aus verschiedenen Datensätzen die Präzision von Vorhersagemodellen erheblich verbessert und eine zuverlässigere Einschätzung der Effizienz von Leit-RNAs ermöglicht. „Die Erweiterung unserer Trainingsdaten durch Zusammenführung mehrerer Experimente ist essenziell, um bessere Vorhersagemodelle zu schaffen. Vor unserer Studie war der Mangel an Daten ein limitierender Faktor für die Vorhersagegenauigkeit“, resümiert Juniorprofessor Barquist. Der jetzt veröffentlichte Ansatz wird künftig sowohl der Biotechnologie als auch der Grundlagenforschung dienen und dabei helfen, effektivere CRISPRi-Experimente zu planen. „Unsere Studie liefert eine Blaupause für die Entwicklung präziserer Instrumente, die die bakterielle Genexpression manipulieren und letztlich dazu beitragen, Krankheitserreger besser zu verstehen und zu bekämpfen“, so Barquist.
Die Ergebnisse im Überblick
Genmerkmale zählen
Die Eigenschaften der Zielgene haben einen erheblichen Einfluss auf die Verringerung der Leit-RNAs in genomweiten Screens.
Datenintegration verbessert Vorhersagen
Die Kombination von Daten aus mehreren CRISPRi-Screens verbessert die Genauigkeit von Vorhersagemodellen erheblich und ermöglicht zuverlässigere Einschätzungen der Effizienz von Leit-RNAs.
Bessere Gestaltung von CRISPRi-Experimenten
Die Studie bietet wertvolle Erkenntnisse für die Gestaltung effektiverer CRISPRi-Experimente, indem sie die Wirksamkeit von Leit-RNAs vorhersagt und so präzise Strategien zur Deaktivierung spezifischer Gene ermöglicht.
Förderung
Die Studie wurde aus Mitteln des Bayerischen Staatsministeriums für Wissenschaft und Kunst durch das Forschungsnetzwerk bayresq.net gefördert.
Originalpublikation
Yu Y, Gawlitt S, Barros de Andrade e Sousa L, Medivan E, Piraud M, Beisel C, Barquist L
Improved prediction of bacterial CRISPRi guide efficiency from depletion screens through mixed-effect machine learning and data integration
Genome Biology (2024), DOI: 10.1186/s13059-023-03153-y
































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/5/4/csm_2024_10_EUREKA__c_HIRI_Luisa_Macharowsky_3_low_01_863006bd0e.jpg)































![[Translate to German:]](/fileadmin/HIRI/__processed__/3/3/csm_2022_07_BEISEL_Chase_2_cropped_Low_a7a341cf03.jpg)





































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/2/8/csm_Collage_WUE_9fa1cd516c.jpg)










![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/0/8/csm_05_Saliba_02_c_HIRI_Luisa_Macharowsky_ohne_Lacoste_low_zugeschnitten_2_01_9822d73a3f.jpg)






![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/c/csm_HIRI-Grundstseinlegung_Foto__c_Mario_Schmitt_79ad64af6d.jpg)













![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/2/csm_2023_04_Smyth_Paper_c_HIRI_Luisa_Macharowsky_2_low_84f993d581.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/e/2/csm_2022_11_HIRI_City_of_Wuerzburg_Child_Care_c_HIRI_Luisa_Macharowsky_2_low_6445b253eb.jpg)
![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/3/9/csm_HIRI_key_visual_CHurtig_606d992d74.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/f/0/csm_Ahsen_blue2_6e28a4ee6b.jpg)












![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/5/csm_2022_02_Marco_Olguin_HIRI-Luisa_Macharowsky_04_low_new_3dc837191c.jpg)


![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/3/c/csm_Joerg_Vogel_Portrait_-_photocredit_Mario_Schmitt__c__HIRI_5013b9643b.jpg)



![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/6/csm_RNA_Delivery_1902376336.jpg)
![[Translate to Deutsch:]](/fileadmin/HIRI/__processed__/7/c/csm_Caliskan_Kibe_Pekarek_105c7d1860.jpg)
![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/c/1/csm_N-Caliskan_M-Zimmer_Zoom_c93c493293.jpg)