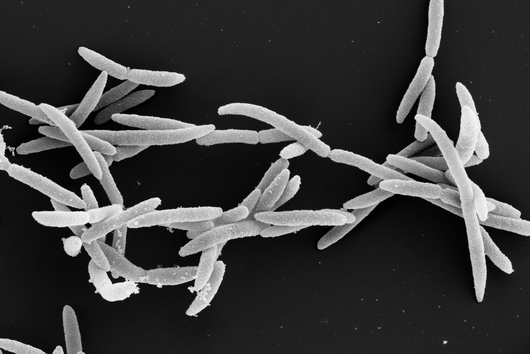
Trypanosomen, die Auslöser der Afrikanischen Schlafkrankheit, tricksen die Immunabwehr ihres Wirts aus, indem sie ihre Oberfläche immer wieder verändern. Ein internationales Forschungsteam, an dem auch Dr. Emmanuel Saliba vom Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) in Würzburg beteiligt war, hat nun das komplette Genom des Parasiten sequenziert und wichtige Aspekte seiner molekularen Strategie aufgeklärt. Über ihre Ergebnisse berichten die Wissenschaftler im Fachmagazin Nature. Das HIRI ist ein Standort des Helmholtz-Zentrums für Infektionsforschung (HZI) in Kooperation mit der Julius-Maximilians-Universität Würzburg.
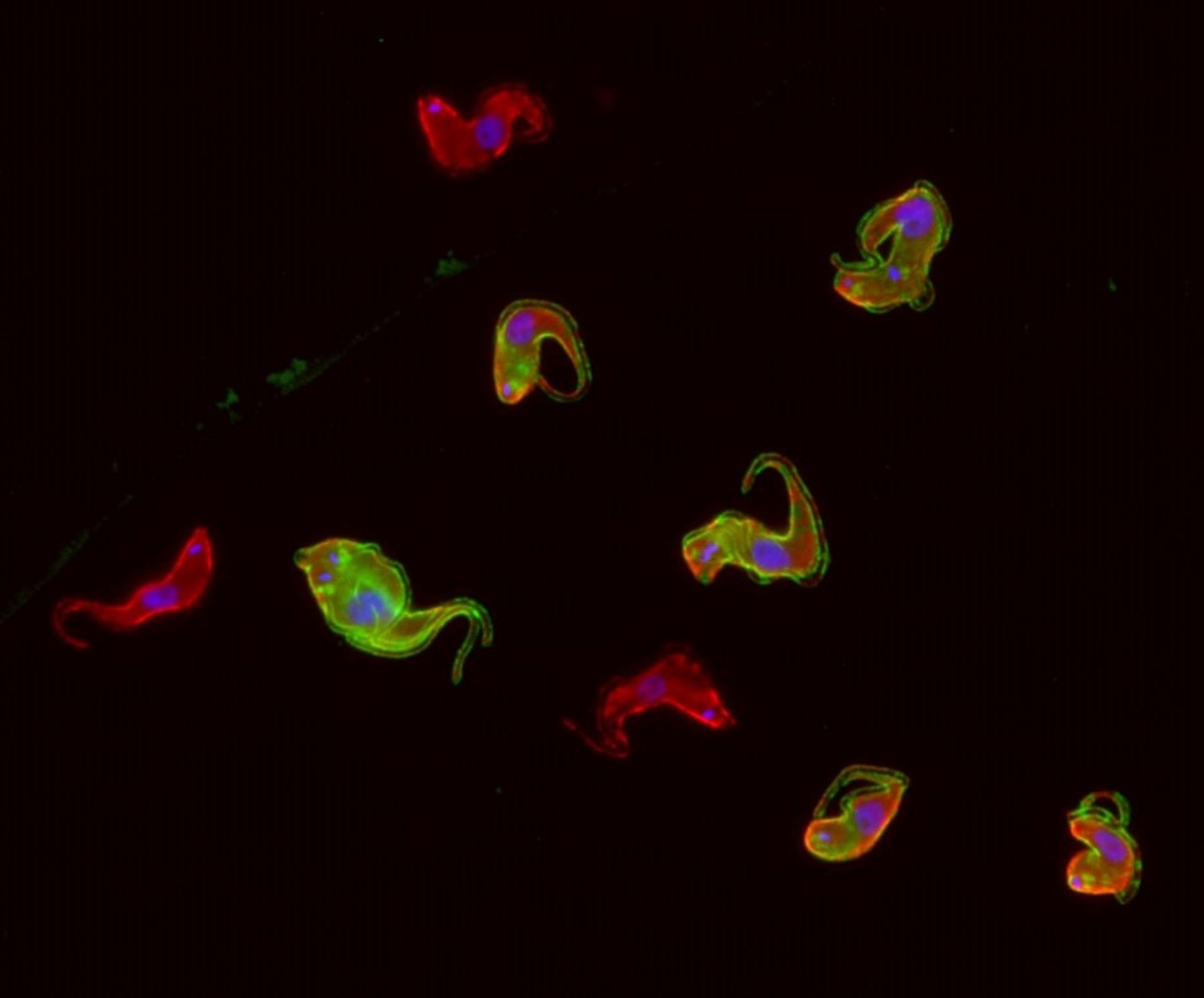
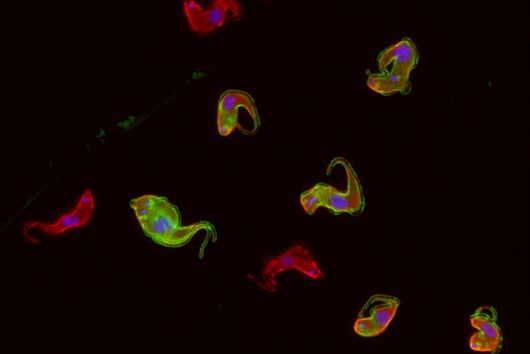
Die einzelligen Parasiten namens Trypanosoma lösen beim Menschen die Afrikanische Schlafkrankheit aus, die tödlich enden kann. Prof. Nicolai Siegel, Leiter der Arbeitsgruppe „Molekulare Parasitologie“ an der Ludwig-Maximilians-Universität München (LMU), erforscht an ihrem Beispiel den ständigen Wettkampf, in dem Parasiten und ihre Wirte stehen: Das Immunsystem des Wirts bekämpft den Parasiten, der wiederum Strategien gegen diese Abwehr entwickelt. Trypanosomen verändern ständig ihre Oberfläche, um vom Immunsystem des Wirts nicht erkannt zu werden. Wissenschaftler der Arbeitsgruppe haben nun in Kooperation mit Kollegen der Universität Würzburg, der Universität Köln, des HIRI in Würzburg sowie Forschern in den USA, Großbritannien und Israel die genetischen Mechanismen dieser Abwehr untersucht. Das Team konnte nachweisen, dass bestimmte DNA-Verpackungsproteine die Struktur der Erbsubstanz des Parasiten beeinflussen.
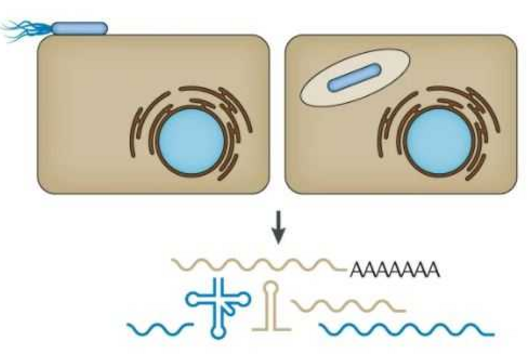
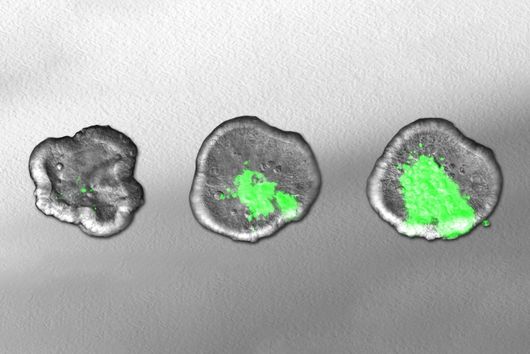
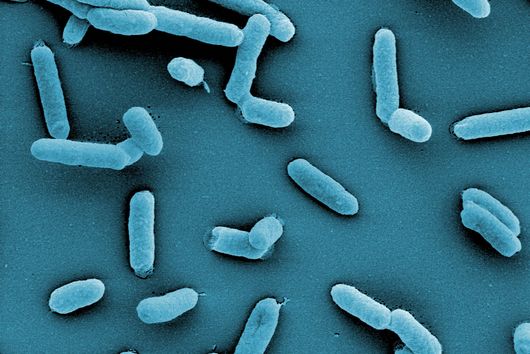
Trypanosomen kommen in verschiedenen Wirbeltieren vor und werden meist von Insekten übertragen. In Säugetieren leben sie vor allem im Blut ihres Wirts. Die von den Wissenschaftlern untersuchte Art Trypanosoma brucei löst nicht nur die Afrikanische Schlafkrankheit aus, sondern ist auch der Erreger der Tierseuche Nagana bei Rindern. Das Genom des Parasiten kodiert für etwa 2000 unterschiedliche Varianten seines Oberflächen-Proteins, wobei nur eine Variante gleichzeitig pro Zelle produziert wird. Da die Immunantwort des Wirts sich immer gegen eine konkrete Variante des Oberflächenproteins richtet, entkommt der Parasit der Immunantwort, indem er das Gen für ein bestimmtes Protein ab- und stattdessen ein anderes anschaltet. Da es einige Zeit dauert, bis die Immunzellen des Wirts das neue Oberflächenprotein als fremd erkennen, erreichen Trypanosomen auf diese Weise eine dauerhafte Infektion.
„Wir interessieren uns vor allem dafür, wie diese genetische Variabilität reguliert wird“, sagt Siegel, dessen Labor zur Tierärztlichen Fakultät gehört und im Biomedizinischen Centrum der LMU angesiedelt ist. Das Erbgut liegt im Zellkern als eng verpackter DNA-Protein-Komplex vor, der als Chromatin bezeichnet wird. Um zu untersuchen, wann welches Gen aktiviert wird, haben die Wissenschaftler erstmals das komplette Genom von T. brucei sequenziert und die dreidimensionale Anordnung der DNA aufgeklärt. Mithilfe eines von Emmanuel Saliba entwickelten Verfahrens zur Einzelzellanalyse des Parasiten konnten sie zeigen, dass der Wechsel zwischen verschiedenen Oberflächenproteinen verstärkt wird, wenn zwei bestimmte Varianten von DNA-Verpackungsproteinen entfernt werden. Diese Varianten befinden sich im Chromatin an denselben Stellen, an denen auch die Gene sitzen, die für die Oberflächenproteine kodieren. Durch die Entfernung dieser Varianten verändert sich zum einen die dreidimensionale Struktur der DNA, und zum anderen lockert sich an den entsprechenden Stellen die ansonsten dicht gepackte DNA, sodass neue Gene zugänglich werden. Beide Effekte gemeinsam ermöglichen neue Interaktionen innerhalb der DNA, sodass andere Gene aktiviert werden. „Entscheidend ist dabei, dass wir beide Proteinvarianten entfernt haben“, betont Siegel. „Fehlt nur eine, ändert sich zwar die dreidimensionale Struktur der DNA, aber es kommt nicht zu einem Switch der Oberflächenproteine.“
Emmanuel Saliba fügt an: „Zum ersten Mal konnten wir das Transkriptom eines einzelnen Trypanosoma-Parasiten sequenzieren und die einzigartige Oberflächenstruktur jedes einzelnen Krankheitserregers entschlüsseln.“ Ein besseres Verständnis dieser Abwehrmechanismen ist auch für die Erforschung anderer Krankheiten wichtig, denn zahlreiche Pathogene haben ähnliche Strategien entwickelt, etwa die Malaria-Erreger, Candida-Pilze und viele Bakterien.
ORIGINALPUBLIKATION:
Laura S. M. Müller, Raúl O. Cosentino, Konrad U. Förstner, Julien Guizetti, Carolin Wedel, Noam Kaplan, Christian J. Janzen, Panagiota Arampatzi, Jörg Vogel, Sascha Steinbiss, Thomas D. Otto, Antoine-Emmanuel Saliba, Robert P. Sebra, T. Nicolai Siegel: Genome organization and DNA accessibility control antigenic variation in trypanosomes. Nature 2018, DOI: 10.1038/s41586-018-0619-8
zum artikel

































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/5/4/csm_2024_10_EUREKA__c_HIRI_Luisa_Macharowsky_3_low_01_863006bd0e.jpg)































![[Translate to German:]](/fileadmin/HIRI/__processed__/3/3/csm_2022_07_BEISEL_Chase_2_cropped_Low_a7a341cf03.jpg)





































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/2/8/csm_Collage_WUE_9fa1cd516c.jpg)










![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/0/8/csm_05_Saliba_02_c_HIRI_Luisa_Macharowsky_ohne_Lacoste_low_zugeschnitten_2_01_9822d73a3f.jpg)






![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/c/csm_HIRI-Grundstseinlegung_Foto__c_Mario_Schmitt_79ad64af6d.jpg)













![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/2/csm_2023_04_Smyth_Paper_c_HIRI_Luisa_Macharowsky_2_low_84f993d581.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/e/2/csm_2022_11_HIRI_City_of_Wuerzburg_Child_Care_c_HIRI_Luisa_Macharowsky_2_low_6445b253eb.jpg)
![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/3/9/csm_HIRI_key_visual_CHurtig_606d992d74.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/f/0/csm_Ahsen_blue2_6e28a4ee6b.jpg)












![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/5/csm_2022_02_Marco_Olguin_HIRI-Luisa_Macharowsky_04_low_new_3dc837191c.jpg)


![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/3/c/csm_Joerg_Vogel_Portrait_-_photocredit_Mario_Schmitt__c__HIRI_5013b9643b.jpg)



![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/6/csm_RNA_Delivery_1902376336.jpg)
![[Translate to Deutsch:]](/fileadmin/HIRI/__processed__/7/c/csm_Caliskan_Kibe_Pekarek_105c7d1860.jpg)
![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/c/1/csm_N-Caliskan_M-Zimmer_Zoom_c93c493293.jpg)