Würzburg, 23. März 2023 – Die Translation von viralen Ribonukleinsäuren (RNAs) wird häufig von nicht-kanonischen Ereignissen angetrieben. Diese sind im viralen Genom kodiert, damit Viren die Kontrolle über die zelluläre Translationsmaschinerie übernehmen können. Eines dieser nicht-kanonischen Ereignisse ist die programmierte ribosomale Leserasterverschiebung. Sie veranlasst das übersetzende Ribosom dazu, ein Nukleotid zurück oder weiter in ein anderes Leseraster zu springen, was die virale Replikation ermöglicht. Forschende des Helmholtz-Instituts Würzburg (HIRI) haben die strukturellen Veränderungen in diesem Prozess mithilfe modernster Technologien entschlüsselt. Ihre Erkenntnisse könnten zur Entdeckung neuer therapeutischer Angriffspunkte beitragen und wurden in der Fachzeitschrift Nucleic Acids Research veröffentlicht.
Translation ist ein essenzieller Prozess in jeder lebenden Zelle und für die Proteinproduktion entscheidend. Viren haben ein Spektrum nicht-kanonischer Ereignisse für jeden Schritt der Translation entwickelt. So können sie die zelluläre Proteinsynthese effizient beeinflussen. Dies ist auch bei SARS-CoV-2 der Fall: Das Virus setzt die ribosomale Leserasterverschiebung (engl.: frameshifting) ein, um die Expression seiner Replikationsenzyme zu ermöglichen.
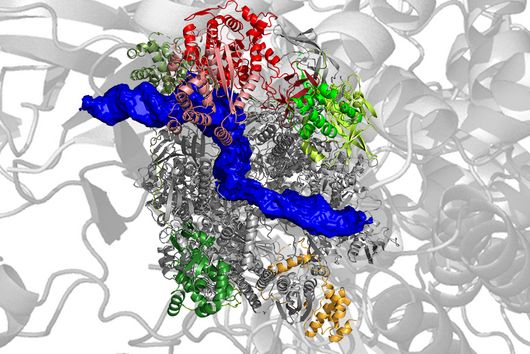
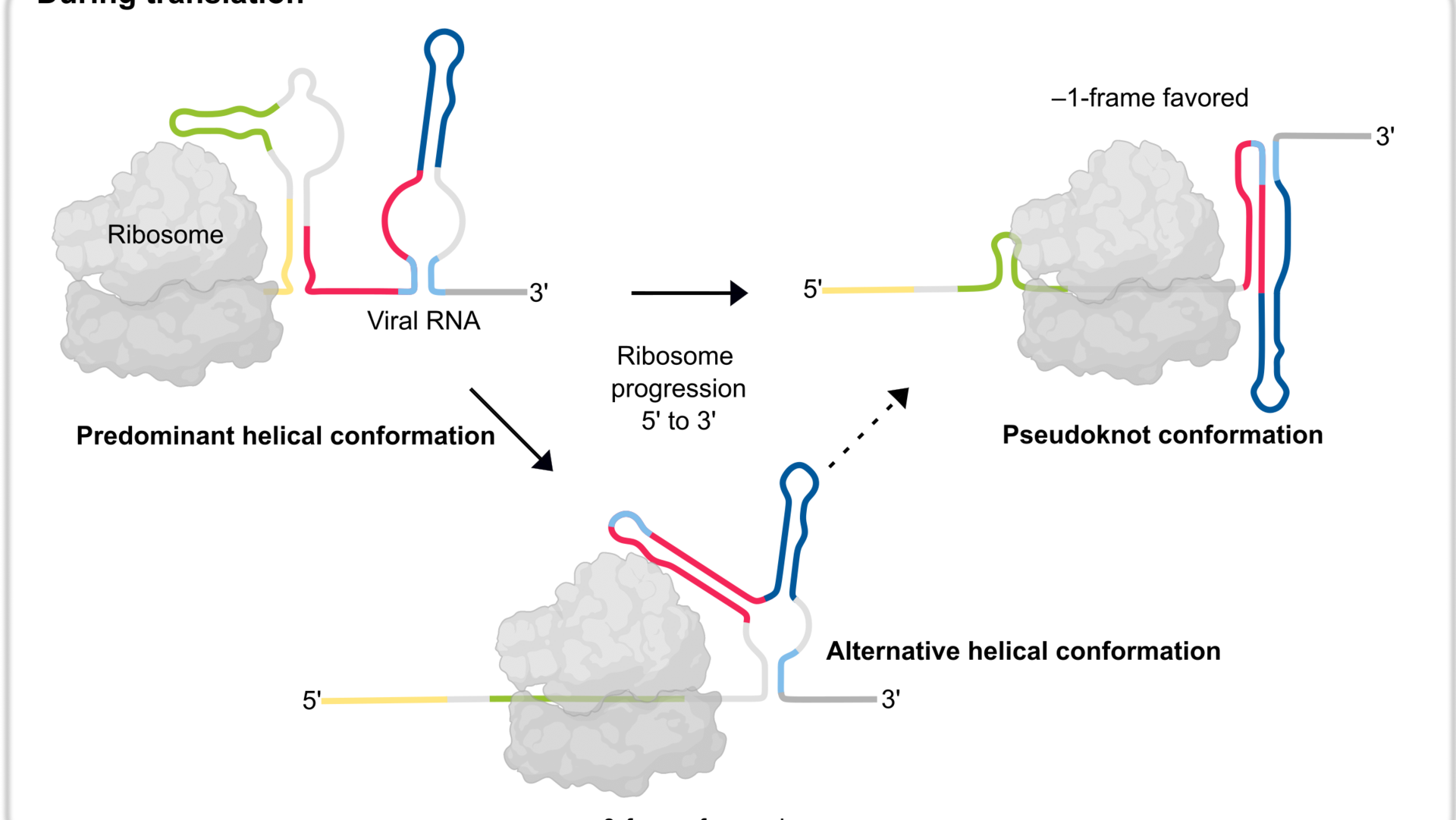
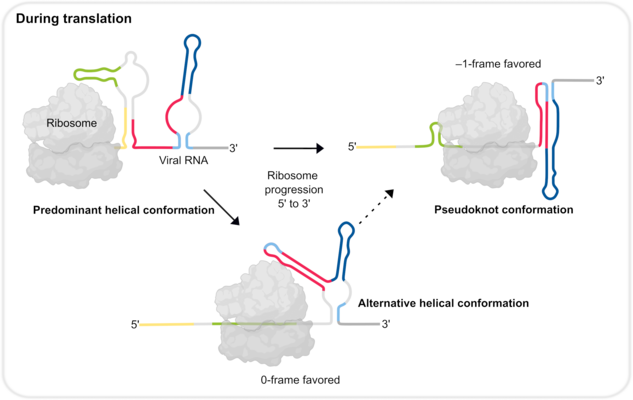
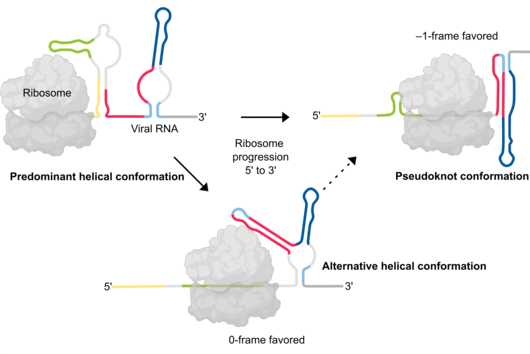
Wie erfolgreich das ist, hängt davon ab, ob bestimmte Teile – Frameshifting-Stimuli – innerhalb der viralen RNA vorhanden sind. Bei einem dieser Teile handelt es sich um eine stabile Struktur, die das Ribosom gewissermaßen stoppt und es in ein alternatives Leseraster zwingt. Die bisherige These für das Pandemievirus SARS-CoV-2 besagt, dass dieses ribosomale Hindernis eine Pseudoknotenstruktur mit einer stabilen RNA-Faltung ist.
Jüngste Versuche der Wissenschaft, diese Pseudoknotenstruktur bei Untersuchungen des vollen Virusgenoms nachzuweisen, schlugen jedoch fehl. Diese scheinbare Inkonsistenz in der Literatur hat die Aufmerksamkeit der Wisenschaftler:innen am Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) in Würzburg erregt, einem Standort des Braunschweiger Helmholtz-Zentrums für Infektionsforschung in Kooperation mit der Julius-Maximilians-Universität (JMU) Würzburg.
Das Virus dekodieren
In ihrer Studie, welche in der Fachzeitschrift Nucleic Acids Research veröffentlicht wurde, konzentrierte sich ein Team von HIRI-Gruppenleiterin Neva Caliskan auf den Teil der viralen RNA, der für die ribosomale Leserasterverschiebung verantwortlich ist. „Unser Ziel war es, die Strategie von SARS-CoV-2 im Detail zu durchdringen,“ berichtet Caliskan. Mithilfe modernster Technologien wie optischer Einzelmolekülpinzetten untersuchten die Forschenden mehrere virale RNA-Varianten, die sich in ihrer Länge unterschieden, sowie einige Mutationen. Auf diese Weise konnten sie die Faltung von RNA-Strukturen in Echtzeit analysieren. Ergänzend zu diesen strukturellen Informationen wurde die Frameshifting-Effizienz bestimmt. Als Gegenprobe untersuchte das Labor von HIRI-Gruppenleiter Redmond Smyth in einem parallelen Ansatz die RNA-Strukturen mittels einer chemischen Methode.
Orchestriertes Zusammenspiel von RNA-Strukturen
Die Studie offenbart, dass ein dynamisches Zusammenspiel von RNA-Strukturen die Effizienz der ribosomalen Leserasterverschiebung – und damit der Vermehrung der Viren – bestimmt. Die Ergebnisse deuten darauf hin, dass die postulierte Pseudoknotenstruktur dabei durchaus essenziell ist. Es zeigte sich jedoch, dass diese besondere Struktur wohl nur vorübergehend während der Translation entsteht. Eine alternative dominante Struktur aus mehreren Helices unterbindet dagegen die Bildung von Pseudoknoten. Während das Ribosom die virale RNA übersetzt, muss es die nachfolgenden RNA-Strukturen sukzessive auffalten. Beim Übersetzen der dominanten Helixform wird der Teil der RNA-Region zu einem bestimmten Zeitpunkt für die Bildung eines Pseudoknoten verfügbar, welcher sich dann zügig formt.
„Mehrere RNA-Strukturen arbeiten bei SARS-CoV-2 zusammen, um die Genexpression ganz fein abzustimmen“, erklärt Lukáš Pekárek, Doktorand im Caliskan-Labor und Erstautor der Studie; „eine Veränderung der Art und Weise, wie sich die Frameshifting-RNA mit benachbarten Regionen der RNA paart, kann die Bildung verschiedener Strukturen und damit die Fähigkeit der Viren beeinflussen, sich in unseren Zellen zu vermehren.“
Wie geht es weiter?
Frameshifting-Stimuli in viralen RNAs sind evolutionär höchst konserviert. Dies macht sie zu einem attraktiven Ziel für die Entwicklung neuer Therapeutika, die robust gegenüber Resistenzen sind. Die Strukturen der Ziel-RNA im Detail zu verstehen, ist daher sehr wichtig. „Bislang konzentrierten sich die meisten Versuche, neue therapeutische Angriffspunkte zu entdecken, auf die Pseudoknoten-Struktur. Obwohl diese Struktur im Frameshifting-Prozess durchaus entscheidend ist, legt unsere Studie nahe, dass sie womöglich kein optimales Ziel für Therapeutika darstellt, da der Pseudoknoten nur sehr kurzlebig ist“, sagt Neva Caliskan. Stattdessen könnte eine Verschiebung des Fokus auf alternative Strukturen eine bessere Trefferquote versprechen, folgern die Wissenschaftler:innen.
Förderung
Die Studie wurde aus Mitteln der Helmholtz-Gemeinschaft und des Europäischen Forschungsrats (ERC) gefördert [948636 an N.C.]; Helmholtz Young Investigator [VH-NG-1347 an R.S.].
Originalpublikation
Pekarek L, Zimmer MM, Gribling-Burrer AS, Buck S, Smyth RP, Caliskan N (2023)
Cis-mediated interactions of the SARS-CoV-2 frameshift RNA alter its conformations and affect function
Nucleic Acids Research, DOI: 10.1093/nar/gkac1184

Pressekontakt

































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/5/4/csm_2024_10_EUREKA__c_HIRI_Luisa_Macharowsky_3_low_01_863006bd0e.jpg)































![[Translate to German:]](/fileadmin/HIRI/__processed__/3/3/csm_2022_07_BEISEL_Chase_2_cropped_Low_a7a341cf03.jpg)





































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/2/8/csm_Collage_WUE_9fa1cd516c.jpg)










![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/0/8/csm_05_Saliba_02_c_HIRI_Luisa_Macharowsky_ohne_Lacoste_low_zugeschnitten_2_01_9822d73a3f.jpg)






![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/c/csm_HIRI-Grundstseinlegung_Foto__c_Mario_Schmitt_79ad64af6d.jpg)













![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/2/csm_2023_04_Smyth_Paper_c_HIRI_Luisa_Macharowsky_2_low_84f993d581.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/e/2/csm_2022_11_HIRI_City_of_Wuerzburg_Child_Care_c_HIRI_Luisa_Macharowsky_2_low_6445b253eb.jpg)
![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/3/9/csm_HIRI_key_visual_CHurtig_606d992d74.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/f/0/csm_Ahsen_blue2_6e28a4ee6b.jpg)












![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/5/csm_2022_02_Marco_Olguin_HIRI-Luisa_Macharowsky_04_low_new_3dc837191c.jpg)


![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/3/c/csm_Joerg_Vogel_Portrait_-_photocredit_Mario_Schmitt__c__HIRI_5013b9643b.jpg)



![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/6/csm_RNA_Delivery_1902376336.jpg)
![[Translate to Deutsch:]](/fileadmin/HIRI/__processed__/7/c/csm_Caliskan_Kibe_Pekarek_105c7d1860.jpg)
![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/c/1/csm_N-Caliskan_M-Zimmer_Zoom_c93c493293.jpg)