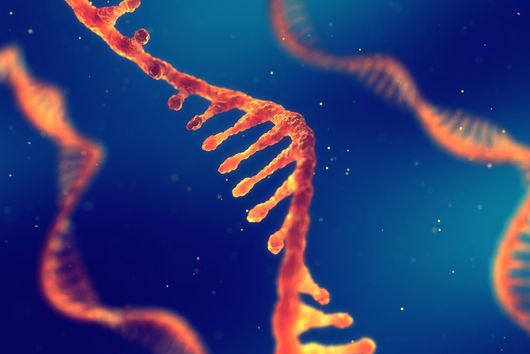
Würzburg, 28. März 2024 – Der Abbau von Ribonukleinsäure (RNA, von engl. ribonucleic acid) ist ein wichtiger Mechanismus, um die Genexpression, also die Umsetzung genetischer Informationen, als Reaktion auf Umweltbelastungen zu steuern. Forschende vom Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) und der Julius-Maximilians-Universität Würzburg (JMU) haben einen statistischen Ansatz entwickelt, mit dem sich die Halbwertszeiten von RNAs in Bakterien genauer als bisher vorhersagen lassen. Mit der neuen Methode konnte das Team feststellen, dass die RNA-Halbwertszeit in Salmonellen dreimal kürzer ist als bisher angenommen. Außerdem konnten sie die Rolle von RNA-bindenden Proteinen beim RNA-Abbau näher beleuchten. Die Ergebnisse sind in der Fachzeitschrift PNAS erschienen.
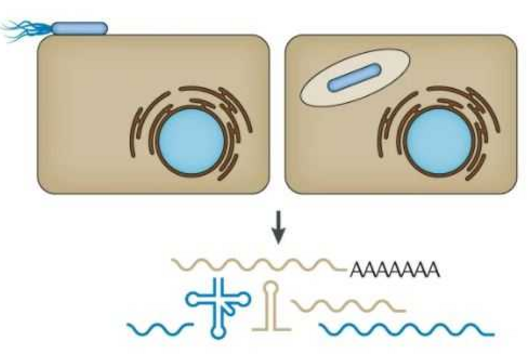
Bakterien benötigen bestimmte Proteine, um sich zu vermehren. Der Bauplan für diese Proteine ist im bakteriellen Erbgut enthalten. Die Proteinbiosynthese erfolgt in zwei Schritten: Zunächst werden bei der Transkription die Informationen, die in der DNA gespeichert sind, in Boten-Ribonukleinsäure (mRNA, von engl. messenger ribonucleic acid) übertragen. Anschließend wird die mRNA im Rahmen der Translation in Proteine übersetzt. Mikroorganismen müssen ihr Proteom, also die Gesamtheit aller Proteine, schnell an sich verändernde Umweltbedingungen anpassen, um ihr Überleben zu sichern. Neben Transkription und Translation ist der RNA-Abbau einer der wichtigsten Prozesse, die die Proteinproduktion steuern.
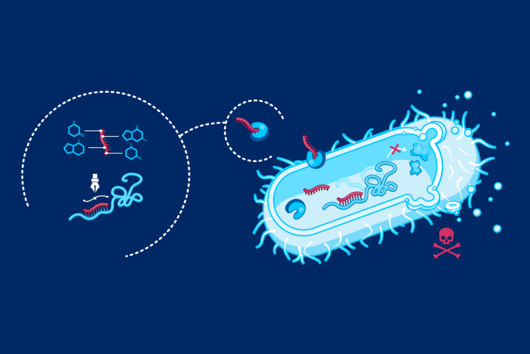
Gängige Methoden zur Bestimmung der Stabilität bakterieller RNA – wie etwa unter Verwendung des Antibiotikums Rifampicin – sind fehleranfällig, was die abgeleiteten Ergebnisse verfälschen kann. Forschende vom Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) und der Julius-Maximilians-Universität Würzburg (JMU) haben einen neuen Ansatz entwickelt, um diese störenden Effekte zu korrigieren. „Wir kombinieren genomische Hochdurchsatzdaten mit einem Bayes'schen Statistikmodell. Dieser Bayes'sche Ansatz ermöglicht es uns, unterschiedliche Annahmen darüber aufzustellen, wie die Datensätze gewonnen wurden. Wir können dann direkt testen, wie gut diese konkurrierenden Hypothesen die tatsächlich beobachteten Daten erklären“, erläutert Lars Barquist. Der Bioinformatiker hat die Studie initiiert und leitet eine Forschungsgruppe am Würzburger Helmholtz-Institut, einem Standort des Braunschweiger Helmholtz-Zentrums für Infektionsforschung (HZI) in Kooperation mit der JMU. Außerdem hat Barquist eine Professur an der kanadischen University of Toronto inne. „Unsere Methode macht die Analyse der RNA-Stabilität unter verschiedenen Bedingungen oder in mutierten Stämmen möglich. Sie stellt ein neues und leistungsfähiges Werkzeug dar, um biologische Parameter aus komplexen Datensätzen zu gewinnen“, so Barquist.
Geringer als erwartet
Mithilfe des neuen Ansatzes haben die Wissenschaftler:innen die Halbwertszeit von RNAs in Salmonella enterica serovar Typhimurium untersucht. Ihr überraschendes Ergebnis: Sie ist etwa dreimal kürzer als bisher angenommen, nämlich knapp eine Minute statt ungefähr drei Minuten. Die Daten der in der Fachzeitschrift Proceedings of the National Academy of Sciences (PNAS) veröffentlichten Studie deuten darauf hin, dass die Halbwertszeiten bakterieller RNAs im Allgemeinen deutlich überschätzt wurden. Sie dürften zu einer generellen Neubewertung der Abbauraten für andere Bakterien führen, da diese in der Regel mit dem hier verwendeten Rifampicin-RNA-Stabilitätstest bestimmt wurden. „RNA-Abbauraten sind einer der grundlegenden Parameter der Genexpression. Unsere Erkenntnisse haben Auswirkungen auf fast alle Studien, die sich mit der Anpassung des Transkriptoms, der Gesamtmenge aller transkribierten Gene, und des Proteoms von Bakterien an verschiedene Umgebungen – wie etwa während einer Infektion oder nach einer Antibiotikabehandlung – befassen“, ordnet Laura Jenniches, Postdoktorandin in der Gruppe von Lars Barquist und Erstautorin der Studie, das Ergebnis ein.
Veränderter Blick auf Proteine
Die neue Methode ermöglichte es dem Forschungsteam außerdem, die Wechselwirkungen zwischen RNA-bindenden Proteinen (RBPs) und dem RNA-Abbau genauer zu untersuchen. Zwar ist bekannt, dass diese Proteine in Bakterien an der post-transkriptionellen Regulation beteiligt sind, ihr Einfluss auf die RNA-Halbwertszeit ist jedoch noch nicht umfassend erforscht. Im Rahmen ihrer Untersuchungen konnten die Forschenden große Transkriptkohorten identifizieren, deren Stabilität sich veränderte, nachdem die RNA-bindenden Proteine ausgeschaltet worden waren. Diese Beobachtungen liefern neue Erkenntnisse über die Rolle der RBPs bei der Gestaltung des Transkriptoms. Sie deuten darauf hin, dass diese Proteine überlappende Rollen bei der Aufrechterhaltung des zellulären Gleichgewichts spielen. „Unsere Methode erlaubt es uns, die Aktivität des RBP-Netzwerks genauer zu untersuchen und so Einblicke zu gewinnen, wie die Proteine Reaktionen auf Stress steuern. Diese Studie ist ein erster Schritt auf dem Weg zu einem besseren Verständnis der durch RBPs koordinierten globalen Regulation in bakteriellen Krankheitserregern“, so Barquist.
Förderung
Die Studie wurde durch das Bayerische Staatsministerium für Wissenschaft und Kunst im Rahmen des Forschungsnetzwerks bayresq.net gefördert.
Originalpublikation
Jenniches L, Michaux C, Popella L, Reichardt S, Vogel J, Westermann AJ, Barquist L
Improved RNA stability estimation through Bayesian modeling reveals most Salmonella transcripts have subminute half-lives
PNAS (2024), DOI: 10.1073/pnas.2308814121




































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/5/4/csm_2024_10_EUREKA__c_HIRI_Luisa_Macharowsky_3_low_01_863006bd0e.jpg)































![[Translate to German:]](/fileadmin/HIRI/__processed__/3/3/csm_2022_07_BEISEL_Chase_2_cropped_Low_a7a341cf03.jpg)





































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/2/8/csm_Collage_WUE_9fa1cd516c.jpg)










![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/0/8/csm_05_Saliba_02_c_HIRI_Luisa_Macharowsky_ohne_Lacoste_low_zugeschnitten_2_01_9822d73a3f.jpg)






![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/c/csm_HIRI-Grundstseinlegung_Foto__c_Mario_Schmitt_79ad64af6d.jpg)













![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/2/csm_2023_04_Smyth_Paper_c_HIRI_Luisa_Macharowsky_2_low_84f993d581.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/e/2/csm_2022_11_HIRI_City_of_Wuerzburg_Child_Care_c_HIRI_Luisa_Macharowsky_2_low_6445b253eb.jpg)
![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/3/9/csm_HIRI_key_visual_CHurtig_606d992d74.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/f/0/csm_Ahsen_blue2_6e28a4ee6b.jpg)












![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/5/csm_2022_02_Marco_Olguin_HIRI-Luisa_Macharowsky_04_low_new_3dc837191c.jpg)


![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/3/c/csm_Joerg_Vogel_Portrait_-_photocredit_Mario_Schmitt__c__HIRI_5013b9643b.jpg)



![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/6/csm_RNA_Delivery_1902376336.jpg)
![[Translate to Deutsch:]](/fileadmin/HIRI/__processed__/7/c/csm_Caliskan_Kibe_Pekarek_105c7d1860.jpg)
![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/c/1/csm_N-Caliskan_M-Zimmer_Zoom_c93c493293.jpg)