Würzburg, 7. Januar 2026 – Die „CRISPR-Genschere“ ist eine bedeutende Grundlage für Technologien zur Genom-Editierung in vielen Bereichen – von Biologie über Medizin bis hin zu Landwirtschaft und Industrie. Dass CRISPR-Cas-Systeme, die ihren Ursprung bilden, noch vielseitiger sind als gedacht, konnte ein Team des Helmholtz-Instituts für RNA-basierte Infektionsforschung (HIRI) in Würzburg nachweisen. Gemeinsam mit dem Braunschweiger Helmholtz-Zentrum für Infektionsforschung (HZI) und der Utah State University (USU) in Logan, USA, haben die Forschenden eine gänzlich neue CRISPR-Abwehrstrategie entdeckt: Im Gegensatz zu bekannten Nukleasen schneidet Cas12a3 spezifisch für die Proteinbildung wichtige Transfer-Ribonukleinsäuren (tRNA, von engl. transfer ribonucleic acid), um infizierte Zellen lahmzulegen. Seine Ergebnisse hat das Team heute im Fachmagazin Nature veröffentlicht.
Bakterien verfügen über eine Vielzahl von Mechanismen, um Eindringlinge wie etwa Viren abzuwehren. Eine dieser Strategien besteht darin, Transfer-Ribonukleinsäuren (tRNA, von engl. transfer ribonucleic acid) zu spalten. Solche tRNAs sind in sämtlichen Zellen vorhanden und spielen eine grundlegende Rolle bei der Übersetzung von Boten-RNA (mRNA, von engl. messenger RNA) in lebenswichtige Proteine. Sie zu deaktivieren, schränkt die Proteinproduktion ein, was dazu führt, dass die infizierte Zelle in einen Ruhezustand übergeht. In der Folge kann sich der Angreifer nicht weiter vermehren und in der Bakterienpopulation ausbreiten.
Eine gängige bakterielle Abwehrmethode, die bisher nicht mit dem Spalten von tRNAs in Verbindung stand, sind CRISPR-Cas-Systeme. CRISPR nutzt RNA-gesteuerte Proteine, sogenannte Cas-Nukleasen (von CRISPR-assoziiert), um Eindringlinge anhand ihres Erbguts zu erkennen und gezielt unschädlich zu machen. Ermitteln sie einen Erreger, führen die Nukleasen eine für jedes System einzigartige Immunantwort aus. Dazu gehört beispielsweise das Schneiden bestimmter Angreifer-DNA oder das Einleiten eines Wachstumsstopps durch umfassenden RNA- und DNA-Abbau. Die Forschung hat sich diese Mechanismen bereits auf vielfältige Weise zunutze gemacht und in CRISPR eine wichtige Grundlage für Technologien zur Genom-Editierung erkannt. Dass CRISPR-Cas im Rahmen einer Immunantwort auch bevorzugt auf tRNAs abzielt, war allerdings nicht bekannt – bis jetzt.
Eine unerwartete Entdeckung
Forschende am Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI), einem Standort des Braunschweiger Helmholtz-Zentrums für Infektionsforschung (HZI) in Kooperation mit der Julius-Maximilians-Universität Würzburg (JMU), haben gemeinsam mit Wissenschaftler:innen des HZI und der Utah State University (USU) einen neuartigen CRISPR-Mechanismus entdeckt, der auf eben diese tRNAs abzielt. „Dieser Fund war gänzlich unerwartet“, sagt Chase Beisel, affiliierter Abteilungsleiter am HIRI und korrespondierender Autor der Studie, die heute im Fachmagazin Nature erschienen ist. „Eigentlich hatte sich unser Team mit Proteinen befasst, die mit einer einzigartigen Nuklease namens Cas12a2 in Verbindung stehen“, fügt Beisel hinzu.
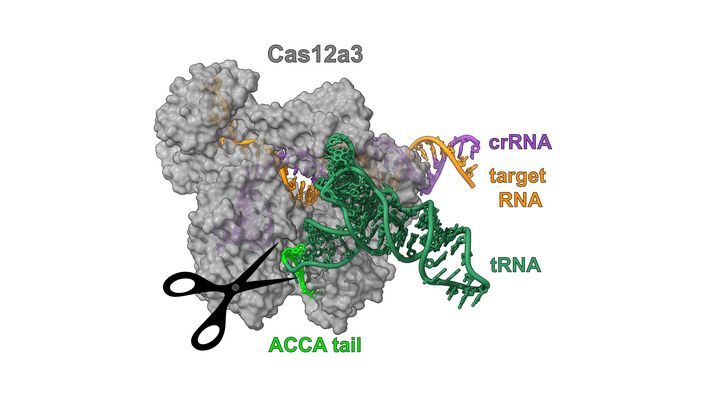
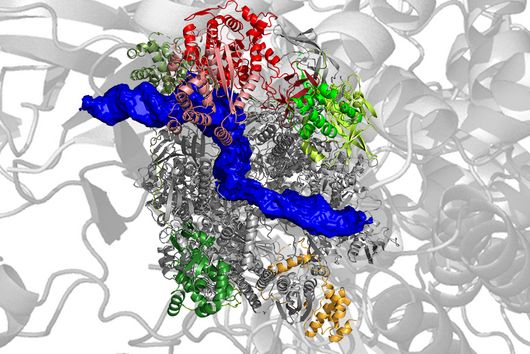
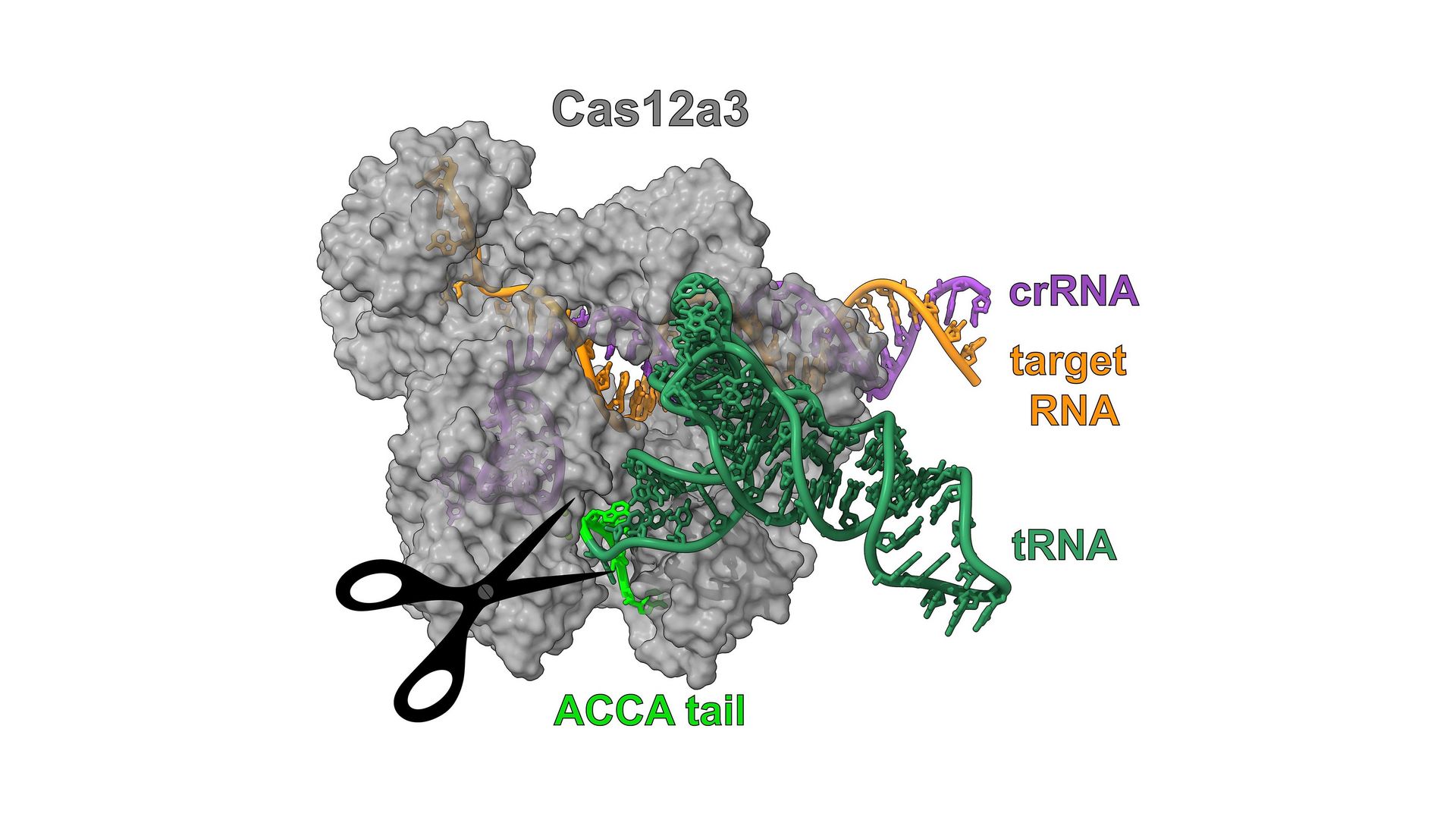
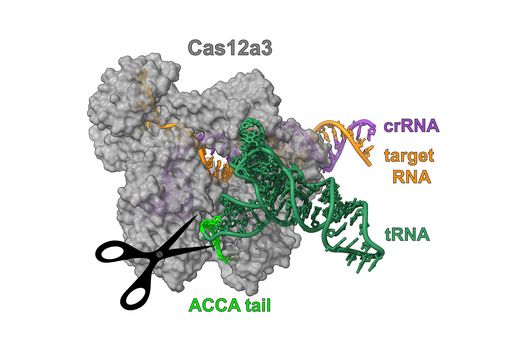
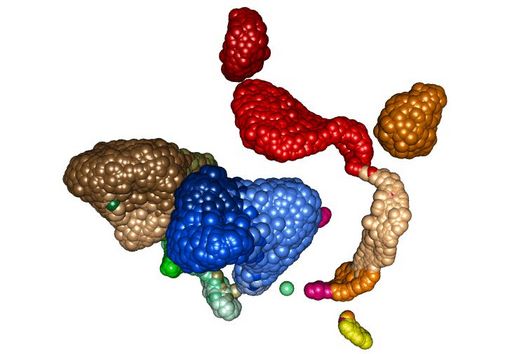
Kryo-Elektronenmikroskopstruktur der Nuklease Cas12a3, die den Schwanz einer Transfer-RNA (tRNA) spaltet. © HZI / Biao Yuan
In zwei im Januar 2023 in der Zeitschrift Nature veröffentlichten Studien beschrieben Teams, denen auch Chase Beisel angehörte, wie sie Cas12a2 in einer Familie von Nukleasen gefunden hatten, die ausschließlich DNA schneiden. Im Gegensatz dazu war Cas12a2 in der Lage, sowohl RNA als auch DNA weitläufig abzubauen. „Wir stellten die Hypothese auf, dass in dieser Proteinfamilie weitere besondere Funktionen verborgen sein könnten. Und wir hatten Recht: So konnten wir Cas12a3 mit seinen einzigartigen Eigenschaften entdecken“, ergänzt Oleg Dmytrenko, ehemaliger Postdoc im Beisel-Labor und Erstautor der jüngst in Nature erschienenen Studie.
Das komplette Gegenteil
Zwar verwendet Cas12a3 wie Cas12a2 ebenfalls RNA, um fremde RNA-Sequenzen ausfindig zu machen. Eine Übereinstimmung führt jedoch dazu, dass Cas12a3 seine Form ändert, sich speziell an eine Schwanzregion der tRNA bindet und sie schneidet. „Diese Genauigkeit macht Cas12a3 zu einem exakten Gegenstück seiner nächsten Verwandten Cas12a2, die eher unspezifisch, aber dafür umfassend schneidet“, sagt Ryan Jackson, Professor an der USU und ebenfalls korrespondierender Autor der Studie.
Beim sogenannten 3‘-Schwanz handelt es sich um den am besten konservierten Teil aller tRNAs. Das bedeutet, dass er über viele Organismen hinweg gleichgeblieben ist. Denn: Diese Region ist entscheidend für die Funktion und Stabilität der RNA und hat sich deswegen evolutionär kaum verändert. An ihr ist eine aktivierte Aminosäure gebunden, der Hauptbaustein von Proteinen. Während der Proteinbildung werden Aminosäuren von der tRNA auf eine wachsende Polypeptidkette übertragen. Daher ist das Entfernen des tRNA-Schwanzes ein wirksames Mittel, um die Herstellung von Proteinen, einem für die Zelle lebenswichtigen Prozess, zu blockieren.
Die Struktur von Cas12a3, die das Team mithilfe von Kryo-Elektronenmikroskopie am HZI beleuchten konnte, zeigt, wie die Nuklease tRNAs erkennt: „Wir konnten einen einzigartigen Teil identifizieren, den wir ‚tRNA-Ladedomäne‘ getauft haben“, erklärt Dirk Heinz, Abteilungsleiter am HZI und weiterer korrespondierender Autor. „Seine Aufgabe besteht darin, den 3′-Schwanz der tRNA präzise so zu positionieren, dass er sich an der richtigen Stelle für die Spaltung befindet“, ergänzt Biao Yuan, Postdoc im Heinz-Labor und ebenfalls Erstautor des Artikels.
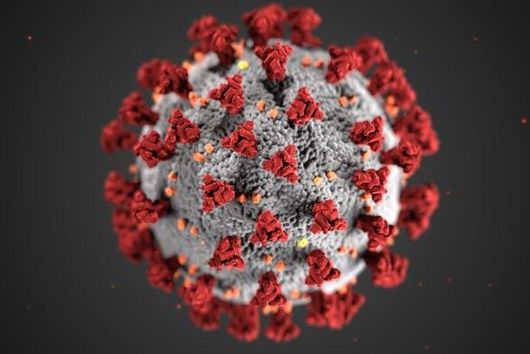
Die hohe Präzision konnten die Forschenden sich direkt zunutze machen: Sie kombinierten Cas12a3 mit zwei weiteren Nukleasen, die ebenfalls zielgenau schneiden, sich jedoch auf andere spezifische RNAs konzentrieren. Mithilfe dieser Kombination war es dem Team möglich, gleichzeitig RNAs von drei verschiedenen Viren – dem Influenzavirus, dem Respiratorischen Syncytial-Virus (RSV) und SARS-CoV-2 – nachzuweisen. „Damit waren wir nicht nur in der Lage, die Grenzen der CRISPR-basierten Diagnostik zu verschieben. Unsere Forschungsergebnisse könnten perspektivisch auch kostengünstige und einfach durchzuführende Point-of-Care-Tests gegen eine Vielzahl von Krankheiten ermöglichen“, sagt Beisel.
Verborgene Vielfalt
Das Spalten von tRNA-Schwänzen stellt eine neue CRISPR-Immunantwort dar und zeugt damit von den vielfältigen Möglichkeiten, mit denen Bakterien Infektionen abwehren können. Die Arbeit rückt somit die enorme funktionelle Vielfalt in den Fokus, die in bereits bekannten bakteriellen Abwehrmechanismen verborgen ist und die es zu untersuchen gilt.
Seine nächsten Schritte hat das Forschungsteam schon geplant: „In der Hoffnung, weitere Variationen zu entdecken, wollen wir diesen winzigen Ausschnitt der CRISPR-Abwehrmechanismen noch tiefergehend erforschen“, blickt Heinz voraus. „Darüber hinaus planen wir, Cas12a3 als Technologie für die molekulare Diagnostik und andere Anwendungen nutzbar zu machen“, schließt Jackson.
Kooperationen
Diese Studie ist das Ergebnis einer Zusammenarbeit zwischen den Laboren von Chase Beisel und Redmond Smyth am Würzburger Helmholtz-Institut für RNA-basierte Infektionsforschung, dem Helmholtz-Zentrum für Infektionsforschung in Braunschweig, der Utah State University in den USA, der Jagiellonen-Universität in Polen und dem Institute of Science and Technology Austria (ISTA) in Österreich.
Förderung
Diese Arbeit wurde durch Fördermittel des Europäischen Forschungsrats (Förderung für Chase Beisel), der Familie R. Gaurth Hansen (Förderung für Ryan Jackson), der National Institutes of Health (Förderung für Ryan Jackson), des „PostDoc Plus“-Programms der Graduiertenschule für Lebenswissenschaften der Julius-Maximilians-Universität Würzburg (Förderung für Oleg Dmytrenko), der Deutschen Forschungsgemeinschaft (DFG) im Rahmen der Exzellenzstrategie Deutschlands – dem Berliner Mathematikforschungszentrum MATH+ (Förderung für Max von Kleist) und der Helmholtz-Gemeinschaft unterstützt.
Originalpublikation
Dmytrenko O, Yuan B, Crosby KT, Krebel M, Chen X, Nowak JS, Chramiec-Głąbik A, Filani B, Gribling-Burrer AS, van der Toorn W, von Kleist M, Achmedov T, Smyth RP, Glatt S, Bravo JPK, Heinz DW, Jackson RN, Beisel CL
RNA-triggered Cas12a3 cleaves tRNA tails to execute bacterial immunity
Nature (2026). DOI: 10.1038/s41586-025-09852-9





![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/b/9/csm_Jahresrueckblick_2025_963859644e.jpg)








![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/e/6/csm_2025_11_20_Neubaufuehrung_fuer_Mitarbeitende_c_HIRI_Yara_Huisl_06_low_0cac9755ff.jpg)
















































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/5/4/csm_2024_10_EUREKA__c_HIRI_Luisa_Macharowsky_3_low_01_863006bd0e.jpg)































![[Translate to German:]](/fileadmin/HIRI/__processed__/3/3/csm_2022_07_BEISEL_Chase_2_cropped_Low_a7a341cf03.jpg)





































![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/2/8/csm_Collage_WUE_9fa1cd516c.jpg)










![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/0/8/csm_05_Saliba_02_c_HIRI_Luisa_Macharowsky_ohne_Lacoste_low_zugeschnitten_2_01_9822d73a3f.jpg)






![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/c/csm_HIRI-Grundstseinlegung_Foto__c_Mario_Schmitt_79ad64af6d.jpg)













![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/c/2/csm_2023_04_Smyth_Paper_c_HIRI_Luisa_Macharowsky_2_low_84f993d581.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/e/2/csm_2022_11_HIRI_City_of_Wuerzburg_Child_Care_c_HIRI_Luisa_Macharowsky_2_low_6445b253eb.jpg)
![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/3/9/csm_HIRI_key_visual_CHurtig_606d992d74.jpg)





























![[Translate to German:] [Translate to German:]](/fileadmin/HIRI/__processed__/f/0/csm_Ahsen_blue2_6e28a4ee6b.jpg)












![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/5/csm_2022_02_Marco_Olguin_HIRI-Luisa_Macharowsky_04_low_new_3dc837191c.jpg)


![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/3/c/csm_Joerg_Vogel_Portrait_-_photocredit_Mario_Schmitt__c__HIRI_5013b9643b.jpg)



![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/a/6/csm_RNA_Delivery_1902376336.jpg)
![[Translate to Deutsch:]](/fileadmin/HIRI/__processed__/7/c/csm_Caliskan_Kibe_Pekarek_105c7d1860.jpg)
![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIRI/__processed__/c/1/csm_N-Caliskan_M-Zimmer_Zoom_c93c493293.jpg)